- Aneuploidien in der Präimplantationsdiagnostik
Embryonale Aneuploidie (Zugewinn oder Verlust ganzer Chromosomen) ist ein häufiges Phänomen sowohl bei natürlich als auch bei in vitro befruchteten Embryonen. Die Aneuploidierate von Embryonen steigt mit zunehmendem mütterlichen Alter, jedoch auch schon bei jungen Frauen scheint diese ein signifikanter Faktor zu sein, der die Reproduktionseffizienz limitiert. NGS (Next-Generation-Sequenzierung) hat sich als die am besten geeignete Technologie für Aneuploidie-Tests in Trophektoderm-Biopsien erwiesen, die zuverlässige Ergebnisse, hohen Durchsatz und Kosteneffizienz bietet. Jüngste Studien deuten darauf hin, dass NGS möglicherweise auch auf verbrauchten Embryokulturmedien für die nicht-invasive Präimplantationsdiagnostik angewendet werden könnte.
L’ aneuploïdie embryonnaire (gain ou perte de chromosomes entiers) est un phénomène courant tant chez les embryons fécondés naturellement que chez ceux fécondés in vitro. Le taux d’ aneuploïdie chez les embryons augmente avec l’ âge maternel, mais même chez les jeunes femmes, ce dernier semble être un facteur important limitant l’ efficacité de la reproduction. Le séquençage de nouvelle génération (NGS) s’est révélé être la technologie la plus appropriée pour le dépistage des aneuploïdies dans les biopsies de trophectoderme, offrant des résultats fiables, au débit élevé avec un bon rapport coût-efficacité. Des études récentes suggèrent que le NGS pourrait également être appliqué aux milieux de culture d’ embryons usagés pour le diagnostic préimplantatoire non invasif.
Einleitung
Daten über in-vitro-Fertilisations-(IVF)-Zyklen zeigen, dass die endgültige kumulative Lebendgeburtenrate pro Eizellenentnahmezyklus von 54,5 % bei jungen Patientinnen auf 13,4 % bei Frauen im Alter von 41-42 Jahren sinkt. Aus diesem Grund ist eines der höchsten Ziele der Reproduktionsmedizin, im Rahmen der IVF Embryonen mit echtem Reproduktionspotenzial zu identifizieren, was sich durch Implantation, normalen Geburtsverlauf und Geburt eines gesunden Kindes zeigt. Traditionelle Ansätze zur Embryoselektion, wie die morphologische Beurteilung des Embryos und die verlängerte Kultur bis zum Blastozystenstadium (Tag 5-6), haben bisher nur einen begrenzten Nutzen gezeigt (1, 2). So selektierte Embryonen kommen bei Patientinnen unter 35 Jahren nur in ca. 45% der Fälle zur Geburt. Bei Frauen über 42 Jahren wird eine Entbindungsrate von etwa 7% beobachtet. Die embryonale Aneuploidie ist hierbei auch schon bei jungen Frauen ein bedeutender Faktor, der die Effizienz der Reproduktion einschränkt. Fetale Aneuploidie ist mit Spontanaborten assoziiert, wobei bis zu 70% der Ersttrimester-Fehlgeburten aneuploid sind. Bei der IVF sind mehr als die Hälfte der Embryonen aneuploid, und die Aneuploidierate des Embryos steigt mit zunehmendem mütterlichen Alter (3). Bei unter 30-jährigen Frauen sind <30% der getesteten Embryonen aneuploid. Die beschriebene Prävalenz steigt auf ca. 90% an, wenn die Frauen Mitte 40 sind (3). Daher sollte die Analyse des chromosomalen Status des Embryos mittels Präimplantationsdiagnostik auf Aneuploidien (PGT-A) die Auswahl des wahrscheinlich kompetentesten Embryos ermöglichen.
Ursprünglich wurde die PGT-A mittels Fluoreszenz in situ Hybridisierung (FISH) durchgeführt. Dieser Ansatz wurde jedoch nach der Veröffentlichung mehrerer randomisierter kontrollierter Studien (RCTs), die deren Nutzen bewerteten, kontrovers diskutiert, da kein positiver Effekt auf die Lebendgeburtenrate nachgewiesen werden konnte. Zudem zeigte sich, dass die PGT-A mittels FISH die Lebendgeburtenrate bei Frauen im fortgeschrittenen mütterlichen Alter (AMA) sogar signifikant senkt (4). Technische Unzulänglichkeiten wie die mögliche embryonale Schädigung durch die Biopsie-Prozedur, die Fehlerrate und die Einschränkungen der FISH-Analyse, zusammen mit einer intrinsischen biologischen Eigenschaft der analysierten Embryonen, nämlich ihrer Mosaik-Natur, begründen die Unwirksamkeit der Methode. Insbesondere erlaubt die FISH-Technik nur die Analyse einer sehr kleinen Anzahl von Chromosomen und die Interpretation an einer einzelnen Zelle kann sehr anspruchsvoll sein. Diese Probleme führten zur Entwicklung neuer diagnostischer Ansätze, die die Analyse mehrerer Zellen ermöglichen und durch Verwendung der vergleichenden Genom-Hybridisierung mit Arrays oder in jüngerer Zeit mittels Next Generation Sequenzierung (NGS) alle 23 Chromosomenpaare abfragen.
NGS-Technologie
Die Sequenzierung der nächsten Generation (Next Generation Sequencing – NGS) ist heute die am häufigsten angewandte Technik für den Nachweis einer Aneuploidie (Präimplantationsdiagnostik auf Aneuploidie – PGT-A) oder einer Aneusomie (Präimplantationsdiagnostik auf strukturelle Rearrangements – PGT-SR) in der Präimplantationsdiagnostik (PID). Im Vergleich zu anderen Methoden bietet NGS mehr potenzielle Vorteile, darunter geringere Kosten, kürzere Testdauer und eine höhere Auflösung für Chromosomenstörungen (5). In NGS-Protokollen wird DNA aus einer einzelnen Blastomere am Tag 3 der Embryonenspaltung oder aus 5-10 Trophektoderm-Zellen im Blastozystenstadium entweder durch selektive DNA-Amplifizierung oder durch Amplifizierung des gesamten Genoms gewonnen. Das Amplifikat wird dann zur Erstellung einer Bibliothek mit kleinen Fragmenten (100 bis 200 bp) verwendet. Auf den Amplifikationsschritt folgt ein Barcoding-Verfahren. Dabei werden die verschiedenen Proben mit eindeutigen Sequenzen (sog. Barcodes) markiert, was die spätere Zuordnung der Sequenzen zum jeweiligen Embryo ermöglicht, nachdem zunächst die Proben für eine oberflächliche Sequenzierung gepoolter DNA gemischt werden (in der Regel Sequenzierung von weniger als 1% des Genoms eines jeden Embryos). Durch diesen Barcoding-Prozess können je nach Sequenzierplattform bis zu 96 Biopsien in einem Sequenzierlauf gepoolt werden, was die Kosten pro sequenziertem Embryo erheblich optimiert. Die Sequenzen oder «Reads» werden dann mit dem aktuellen menschlichen Referenzgenom abgestimmt. Die relative Anzahl der Fragmente von DNA-Sequenz-Reads, die auf jede Chromosomenregion abgebildet werden, liefert einen Hinweis auf die Ploidie bzw. Somie. So ist z. B. eine Trisomie oder eine segmentale Duplikation mit einer Zunahme der relativen Anzahl von Reads für die betroffene chromosomale Region verbunden, während eine Monosomie oder segmentale Deletion zu einer Abnahme führt (6-9) (Abb. 1).
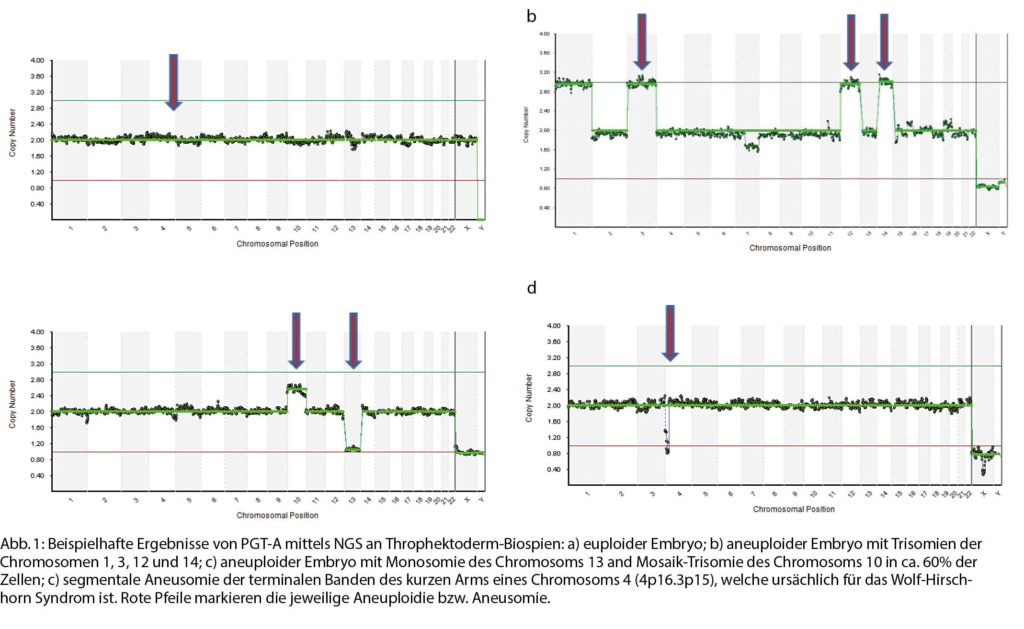
Der klinische Nutzen der PGT-A für alle Chromosomen wurde in einer Reihe von RCTs getestet. Unter diesen konnte eine kürzlich durchgeführte multinationale, multizentrische RCT keinen klinischen Nutzen durch den Einsatz von PGT-A zeigen (10), während andere über eindeutige Vorteile berichteten, wie z. B. erhöhte Implantationsraten pro übertragenem Embryo (ET), verkürzte Zeit bis zum Eintreten einer Schwangerschaft, verringerte klinische Fehlgeburtenraten und Kosteneffizienz, wenn mehr als ein Embryo gewonnen wird (1, 11). Bei Frauen mit AMA zeigte eine aktuelle multizentrische, randomisierte Studie, dass die PGT-A-Gruppe signifikant weniger ETs (68,0 % vs. 90,5% bei den Kontrollen) und geringere Fehlgeburtenraten (2,7% vs. 39,0 % bei den Kontrollen) aufwies. Die Entbindungsrate nach dem ersten Transferversuch war in der PGT-A-Gruppe pro Transfer (52,9% vs. 24,2%) und pro Patientin (36,0% vs. 21,9%) signifikant höher. Bei den kumulativen Entbindungsraten pro Patientin wurden jedoch 6 Monate nach Abschluss der Studie keine signifikanten Unterschiede festgestellt. Allerdings war die mittlere Anzahl der erforderlichen ETs pro Lebendgeburt in der PGT-A-Gruppe im Vergleich zur Kontrollgruppe geringer (1,8 vs. 3,7), ebenso die Zeit bis zur Schwangerschaft (7,7 vs. 14,9 Wochen) (12). In Übereinstimmung mit den Richtlinien der American Society of Reproductive Medicine kann die Verwendung von PGT-A auch einen einzelnen ET ermöglichen (13), ohne die Schwangerschaftsraten zu beeinträchtigen (1), wobei die mit Mehrlingsschwangerschaften verbundenen Komplikationen vermieden werden. Darüber hinaus scheint sich die Trophektoderm-Biopsie weder negativ auf die Implantation auszuwirken (14), noch zu negativen Auswirkungen auf das Neugeborene zu führen (15).
NGS an Trophektoderm-Biopsien ermöglicht auch einen präziseren Nachweis von Aneuploidien in Mosaikform als bisherige Technologien. Embryonaler Mosaizismus ist ein Phänomen, das durch das Vorhandensein von zwei oder mehr genetisch unterschiedlichen Zelllinien gekennzeichnet ist, von denen typischerweise eine eine Chromosomenanomalie aufweist und die andere eine normale chromosomale Konstitution zeigt (16). Obwohl seine Auswirkungen auf die Implantation und das Entwicklungspotenzial von Embryonen nicht vollständig bekannt sind, kann man davon ausgehen, dass Mosaizismus wahrscheinlich die Implantationsrate beeinflusst. Ein solches Phänomen ist bei menschlichen Präimplantationsembryonen relativ häufig und betrifft 15%-90% der Embryonen im Spaltstadium und 30%-40% der Embryonen im Blastozystenstadium (16). Technisch gesehen ist es wichtig, jede NGS-Plattform für die genaue Erkennung von niedriggradigem Mosaizismus zu validieren, um diesen von experimentellem Rauschen zu unterscheiden, was mit der Qualität und Quantität der biologischen Proben und Amplifikationsartefakten zusammenhängt. Zusätzlich bleibt es aufgrund technischer und biologischer Einschränkungen eine Herausforderung, die tatsächliche Inzidenz von Mosaizismus in Präimplantationsembryonen zu erheben. Interessanterweise haben neuere Studien den Ansatz vorgeschlagen, einige Arten von Mosaik-Embryonen nach entsprechender Patientenberatung zu transferieren. Wie erwartet, zeigen die meisten eine geringere Implantations- und höhere Fehlgeburtsrate als beim Transfer euploider Embryonen. Mosaik-Embryonen mit hohem Aneuploidieanteil (≥50%) zeigten hierbei eine signifikant niedrigere klinische Schwangerschaftsrate/ET (15,2% vs. 46,4%), Implantationsrate (24,4% vs. 54,6%) und Lebendgeburtsrate (15,2% vs. 46,6%) als euploide Blastozysten. Im Gegensatz dazu haben Embryonen mit einem geringeren Aneuploidieanteil (< 50%) ein ähnliches klinisches Ergebnis wie euploide Embryonen (17-19).
Bemerkenswert ist in diesem Zusammenhang die kürzlich veröffentlichte Geburt eines anscheinend gesunden Mädchens in der 37. Schwangerschaftswoche mit echtem Mosaizismus, der aus einem bekannten Mosaik-Embryotransfer (35% Monosomie 2-Mosaik) resultierte. Der Fötus zeigte pränatal ein normales Wachstum und keine Auffälligkeiten in der detaillierten Ultraschalluntersuchung. Bei der Geburt zeigte die Chromosomenanalyse des peripheren Blutes eine 2%ige Mosaik-Monosomie 2 (20). Die weitere Entwicklungskontrolle dieses Kindes wird wichtige Informationen hinsichtlich einer möglichen kognitiven Einschränkung liefern.
Zukünftige Entwicklungen
Um embryonales genetisches Material für die Analyse zu gewinnen, ist bislang eine Biopsie erforderlich, bei der eine oder mehrere Zellen (normalerweise 5-10 Zellen) aus dem Embryo bzw. dem Throphektoderm entnommen werden. Diese invasive Prozedur erhöht die Kosten der PGT erheblich und es gab Bedenken, dass die Lebensfähigkeit der Embryonen in einigen Fällen beeinträchtigt werden könnte. Die jüngste Entdeckung von embryonaler DNA in der Blastozelenflüssigkeit (BF) von Blastozysten und in verbrauchten Embryokulturmedien (SCM) hat das Interesse an der Entwicklung von nicht-invasiven Methoden der PGT (niPGT) geweckt, insbesondere für den Nachweis von Aneuploidien und segmentalen Aneusomien (niPGT-A bzw. niPGT-SR). Erste Studien haben einige Erfolge erzielt und deuten darauf hin, dass DNA aus BF- und SCM-Proben für genetische Analysen geeignet sein könnte und somit ein potenzielles Ausgangsmaterial für die PGT darstellt. Dieser Ansatz wäre kostengünstiger als herkömmliche Methoden und würde weniger Fähigkeiten zur Mikromanipulation erfordern, sowie ein geringeres Risiko für die Embryonen darstellen (21). Meist war die diagnostische Effizienz bislang durch technische Komplikationen im Zusammenhang mit der geringen Quantität und Qualität der DNA eingeschränkt. Die berichteten Übereinstimmungen der Ploidie zwischen SCM/BF-Proben und biopsierten embryonalen Zellen variieren stark. In einigen Fällen kann diese Diskrepanz auf embryonalen Mosaizismus oder DNA-Kontamination (meist mütterlichen Ursprungs) zurückzuführen sein. Einige Untersuchungen deuten darauf hin, dass Aneuploidie-Zellen bevorzugt aus dem Embryo eliminiert werden, was dazu führen könnte, dass ihre DNA in SCM- und BF-Proben überrepräsentiert wäre; diese Hypothese erfordert jedoch weitere Untersuchungen (22). Entscheidend ist auch, dass die Protokolle für DNA-Isolierung und Amplifikation optimiert werden, um reproduzierbar eine genaue klinische Diagnose zu erhalten und gleichzeitig den Einfluss von Störfaktoren wie Kontaminationen zu minimieren (22). Weitere Untersuchungen sind erforderlich, um die Mechanismen zu verstehen, die der Freisetzung von embryonaler DNA zugrunde liegen, und um festzustellen, inwieweit dieses Material den wahren genetischen Status des entsprechenden Embryos widerspiegelt (22).
Copyright bei Aerzteverlag medinfo AG
Spezialistin medizinisch-genetische Analytik FAMH
Universität Zürich, Institut für Medizinische Genetik
Wagistrasse 12
8952 Zürich
oneda@medgen.uzh.ch
Die Autorin hat im Zusammenhang mit diesem Artikel keine Interessenskonflikte deklariert.
◆ Die NGS hat sich zur geeignetsten Technologie für Aneuploidie-Tests in Trophektoderm-Biopsien entwickelt, die genaue Ergebnisse mit hohem Durchsatz und klinischem Nutzen liefert. Kürzlich wurde die Kosteneffizienz für die «Baby-Take-Home-Rate» analysiert und die Kosten werden in der Tat nicht mehr als Einschränkung für die Implementierung von Aneuploidie-Tests bei Embryonen angesehen (12).
Messages à retenir
◆ Le NGS est devenu la technologie la plus appropriée pour chercher des aneuploïdies dans des biopsies de trophectoderme, fournissant avec un haut débit des résultats précis, utiles pour la clinique. Récemment, le rapport coût-efficacité a été analysé en se référant au taux de naissances avec enfant vivant (« Take Home Baby Rate »). Il en ressort que le coût n’ est effectivement plus à considérer comme limitatif pour l’ implémentation d’ aneuploïdies chez les embryons (12).
1. Forman EJ, et al. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertil Steril 2013;100(1):100-7 e1.
2. Capalbo A, et al. Correlation between standard blastocyst morphology, euploidy and implantation: an observational study in two centers involving 956 screened blastocysts. Hum Reprod 2014;29(6):1173-81.
3. Franasiak JM, et al. The nature of aneuploidy with increasing age of the female partner: a review of 15,169 consecutive trophectoderm biopsies evaluated with comprehensive chromosomal screening. Fertil Steril 2014;101(3):656-63 e1.
4. Mastenbroek S, et al. Preimplantation genetic screening: a systematic review and meta-analysis of RCTs. Hum Reprod Update 2011;17(4):454-66.
5. Wells D, et al. Clinical utilisation of a rapid low-pass whole genome sequencing technique for the diagnosis of aneuploidy in human embryos prior to implantation. J Med Genet 2014;51(8):553-62.
6. Kung A, et al. Validation of next-generation sequencing for comprehensive chromosome screening of embryos. Reprod Biomed Online 2015;31(6):760-9.
7. Fiorentino F, et al. Development and validation of a next-generation sequencing-based protocol for 24-chromosome aneuploidy screening of embryos. Fertil Steril 2014;101(5):1375-82.
8. Huang J, et al. Validation of a next-generation sequencing-based protocol for 24-chromosome aneuploidy screening of blastocysts. Fertil Steril 2016;105(6):1532-6.
9. Vera-Rodriguez M, et al. Distribution patterns of segmental aneuploidies in human blastocysts identified by next-generation sequencing. Fertil Steril 2016;105(4):1047-55 e2.
10. Munne S, et al. Preimplantation genetic testing for aneuploidy versus morphology as selection criteria for single frozen-thawed embryo transfer in good-prognosis patients: a multicenter randomized clinical trial. Fertil Steril 2019;112(6):1071-9 e7.
11. Neal SA, et al. Preimplantation genetic testing for aneuploidy is cost-effective, shortens treatment time, and reduces the risk of failed embryo transfer and clinical miscarriage. Fertil Steril 2018;110(5):896-904.
12. Rubio C, et al. In vitro fertilization with preimplantation genetic diagnosis for aneuploidies in advanced maternal age: a randomized, controlled study. Fertil Steril 2017;107(5):1122-9.
13. Practice Committee of the American Society for Reproductive Medicine. Electronic address Aao, et al. Guidance on the limits to the number of embryos to transfer: a committee opinion. Fertil Steril 2017;107(4):901-3.
14. Tiegs AW, et al. A multicenter, prospective, blinded, nonselection study evaluating the predictive value of an aneuploid diagnosis using a targeted next-generation sequencing-based preimplantation genetic testing for aneuploidy assay and impact of biopsy. Fertil Steril 2021;115(3):627-37.
15 He H, et al. Neonatal outcomes of live births after blastocyst biopsy in preimplantation genetic testing cycles: a follow-up of 1,721 children. Fertil Steril 2019;112(1):82-8.
16. Munne S, et al. Chromosome mosaicism in human embryos. Biol Reprod 1994;51(3):373-9.
17. Greco E, et al. Healthy Babies after Intrauterine Transfer of Mosaic Aneuploid Blastocysts. N Engl J Med 2015;373(21):2089-90.
18. Fragouli E, et al. Analysis of implantation and ongoing pregnancy rates following the transfer of mosaic diploid-aneuploid blastocysts. Hum Genet 2017;136(7):805-19.
19. Spinella F, et al. Extent of chromosomal mosaicism influences the clinical outcome of in vitro fertilization treatments. Fertil Steril 2018;109(1):77-83.
20. Kahraman S, et al. The birth of a baby with mosaicism resulting from a known mosaic embryo transfer: a case report. Hum Reprod 2020;35(3):727-33.
21. Rubio C, et al. Clinical application of embryo aneuploidy testing by next-generation sequencing. Biol Reprod 2019;101(6):1083-90.
22. Leaver M, et al. Non-invasive preimplantation genetic testing (niPGT): the next revolution in reproductive genetics? Hum Reprod Update 2020;26(1):16-42.
info@gynäkologie
- Vol. 11
- Ausgabe 4
- August 2021






