- Der anspruchsvolle Weg von Genanalyse bis Therapie
Das molekulare Tumorboard ist eine organübergreifende, interdisziplinäre Tumorkonferenz. Das Board hat die Aufgabe, zusätzliche zielgerichtete Therapiemöglichkeiten für Patient*innen nach Ausschöpfung der leitliniengerechten Behandlung aufzuzeigen. Hierzu werden molekularpathologische Untersuchungen auf mögliche prädiktive Biomarker analysiert. Die klinische Klassifikation der Mutationen hilft, rationale, evidenzbasierte, molekular-pathologisch gestützte Behandlungsentscheidungen abzugeben.
The molecular tumourboard is a non-organ-specific, interdisciplinary tumour conference. The board has the task of identifying additional targeted therapy options for patients after guideline-based treatments have been exhausted. For this purpose, molecular pathological examinations are analysed for possible predictive biomarkers. The clinical classification of mutations helps to make rational, evidence-based, molecular pathologysupported treatment decisions.
Key Words: molekulares Tumorboard, ESCAT, Variante, Mutation, Klassifikation
Seit Anfang der 2000er Jahre hat die Onkologie ausserordentliche Veränderungen erfahren. Die Entschlüsselung der menschlichen Genomsequenzen, deren erster Entwurf im Jahr 2001 veröffentlicht wurde, gab der Forschung einen wichtigen Impuls. Seitdem wurden viele Gene, Proteine und Signalwege identifiziert, die an der Krebsentwicklung beteiligt sind. Die Fortschritte beim Verständnis der Krebsbiologie und die technologischen Innovationen erleichtern und beschleunigen fortlaufend die Suche nach Mutationen. Heutzutage können wir mit dem «next generation sequencing» (NGS) 20 bis 500 Gene gleichzeitig untersuchen, die auf Grund ihrer diagnostischen, therapeutischen und prognostischen Bedeutung ausgewählt wurden. Die molekularpathologischen Untersuchungen dienen in erster Linie dazu, prädiktive Biomarker für zugelassene Therapien nachzuweisen. Es ist jedoch unvermeidbar, dass man auch mit Varianten konfrontiert wird, die zwar als prädiktive Biomarker vielversprechend gelten, jedoch für eine Zulassung noch nicht genügend geprüft sind. Es kann zudem vorkommen, dass eine Mutation an Tumorzellen auf eine zugrundeliegende Keimbahnmutation hinweist. Je nach Literatur werde bei der NGS-Untersuchung von Tumoren bei bis zu 15% der Patienten solche pathogene Keimbahnvarianten gefunden (1,2). Die Bestätigung einer hereditären Veranlagung erfolgt dann separat in der Sprechstunde für genetische Beratung.
Es entsteht somit eine grosse Menge an Daten, die auf ihre prädiktive Bedeutung interpretiert werden muss.
Das molekulare Tumorboard
Um der Komplexität der Bedeutung molekularbiologischer Veränderungen gerecht zu werden, haben die University of Michigan (USA) vor etwa 10 Jahren und danach verschiedene universitäre Kliniken weltweit das molekulare Tumorboard (MTB) eingeführt. Das Ziel des MTB ist, auf der Grundlage von molekular-pathologischen Untersuchungen alle potenziellen therapeutischen Strategien zu erkennen und zu diskutieren, die eine zusätzliche Option für den Patienten sein könnten (3,4).
Das MTB besteht aus einem fachärztlichen Kern aus Onkologen und Molekularpathologen. Dazu können auch Genetiker, medizinische Biologen und Bioinformatiker anwesend sein, wobei die personelle Zusammensetzung von Klinik zu Klinik sehr unterschiedlich sein kann. Am häufigsten werden Daten aus NGS-Analysen besprochen.Aber auch Resultate aus Whole Genome Sequencing (WGS), Whole Exome Sequencing (WES), RNA-Sequenzierung, Sanger Sequenzierung, Array Comparative Genomic Hybridization (aCGH), Immunhistochemie und Fluoreszenz-in-situ-Hybridisierung (FISH) werden in die Diskussion miteinbezogen (3). Da ein internationaler Konsens für die Datenauswertung noch aussteht, stützen sich Interpretation und Empfehlung auf lokale Protokolle, sowie Anwendung von Datenbank- und Literaturrecherchen. Die akribische molekulare Charakterisierung der genomischen Aberrationen und die Kenntnisse über die Zuverlässigkeit der Evidenz der klinischen Studien sind unerlässlich für die therapeutische Entscheidung.
Nomenklatur und funktionelle Klassifikation einer Variante
Die Identifikation von Varianten aus einer NGS-basierten Untersuchung wird heutzutage durch Anwendung von Softwares erleichtert. Jede Variante wird in Bezug auf ihre Genposition und Auswirkung auf die Aminosäuresequenz beschrieben. Die Annotation erfolgt basierend auf einer einheitlichen Standard-Nomenklatur nach Vorgabe der Human Genome Variation Society (HGVS) (5).
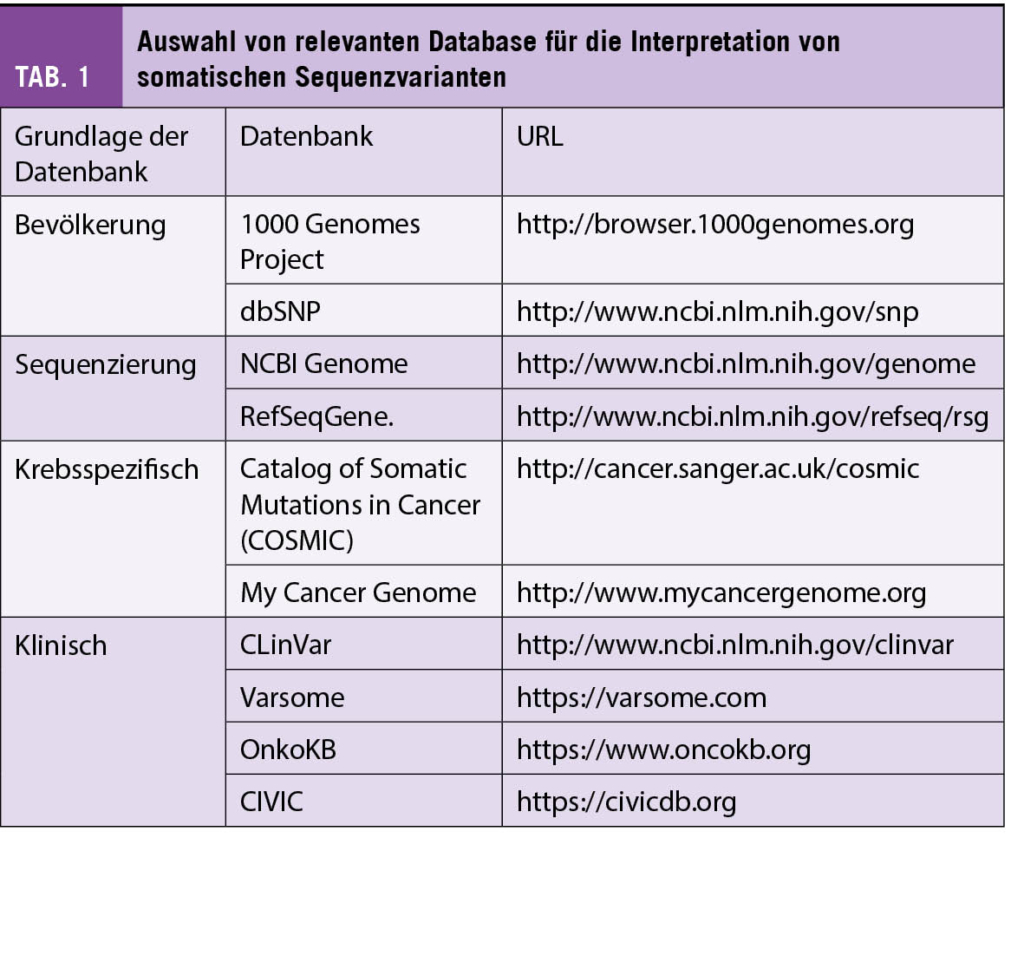
Die Auswirkung einer Mutation auf die biologischen und funktionellen Eigenschaften eines Proteins bestimmt die Pathogenität. Bei der Auswertung der Pathogenität werden unter anderem Kriterien wie Häufigkeit der Mutation in der Population, ihre evolutionäre Konservierung, ihr Einfluss auf die Proteinstruktur und –funktion berücksichtigt. Für die Analyse stehen eine Vielfalt an öffentlichen, privaten oder kommerziellen Databanken und «In-silico»-Tools zur Verfügung (Tab. 1). Der Einbezug eines Bioinformatikers ist allgemein empfohlen, aber vor allem bei der Auswertung von komplexeren Varianten indiziert. Basierend auf den Ergebnissen erfolgt die Einteilung nach der international anerkannten American College of Medical Genetics and Genomics (ACMG) Klassifikation in (i) pathogen, (ii) wahrscheinlich pathogen, (iii) ungewisse Bedeutung, (iv) wahrscheinlich gutartig, oder (v) gutartig (5, 6). Lediglich die Varianten die als pathogen oder wahrscheinlich pathogen klassifiziert werden, sind auch als therapierelevant einzuschätzen. Solche Varianten müssten in jedem molekular-pathologischen Bericht aufgeführt werden.
Klassifikation einer Variante nach der klinischen Evidenz
Die funktionelle Rolle eines Proteins wird durch unzählige Einflüsse reguliert, die sehr gewebespezifisch sind. Deshalb ist die prognostische und prädiktive Relevanz einer Variante von Tumor zu Tumor sehr unterschiedlich und sie wird ausschliesslich von den publizierten genomisch-gestützten Studien bestimmt. Biomarker-assoziierte Therapien mit einem statistisch signifikanten und klinisch relevanten Überlebensvorteil in einer klinischen Studie gelten meistens als «standard of care» und werden früh in der Behandlung eingesetzt. Für alle anderen molekularen Biomarker mit einer schwächeren Datenlage gibt es keine Richtlinien für die optimale Behandlungssequenz.
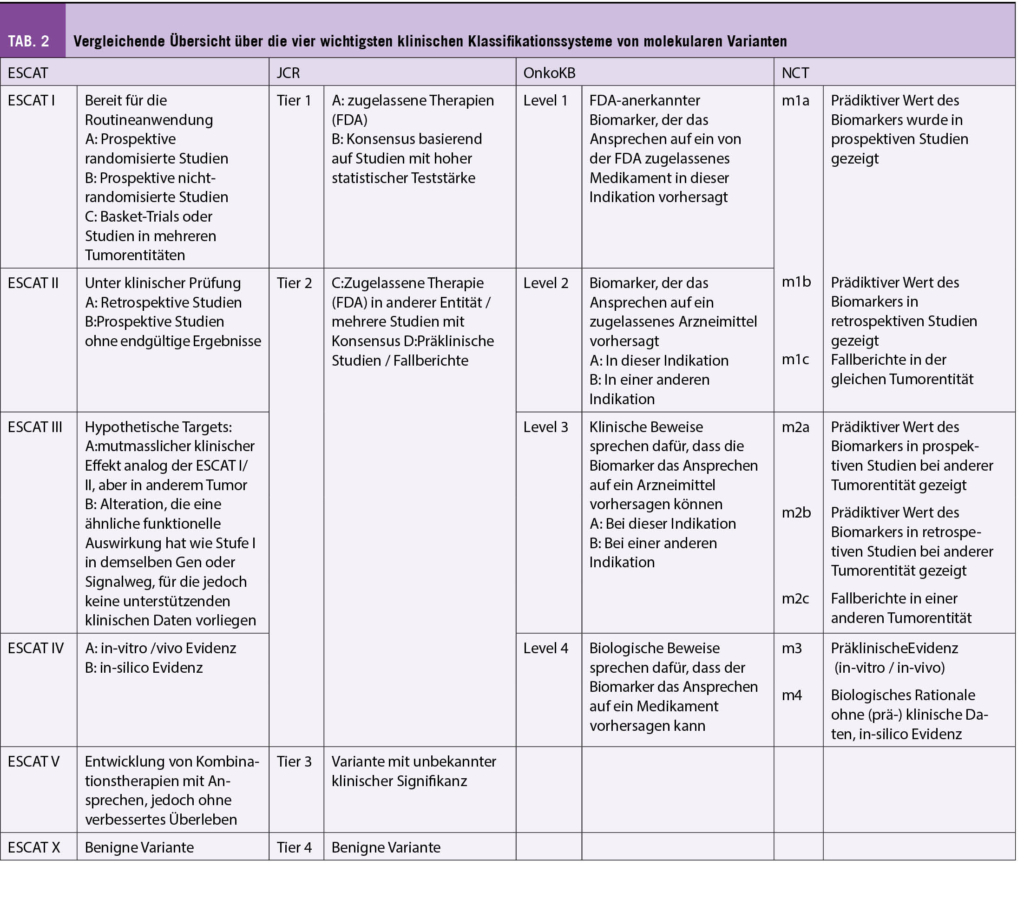
Heutzutage stehen diverse Klassifikationen für die klinische Interpretation von somatischen Varianten zur Verfügung: sie klassifizieren die Varianten in Abhängigkeit der wissenschaftlichen Evidenz und des potentiell klinischen Nutzens. Das erleichtert die Datainterpretation und unterstützt die Therapieauswahl, indem diejenige mit der höchsten Evidenzlage bevorzugt wird. Zu den bekanntesten Klassifikationen zählen:
- Joint Consensus Recommendation (JCR) der Association for Molecular Pathology (AMP), des American College of Medical Genetics and Genomics (ACMG), der American Society of Clinical Oncology (ASCO) und des College of American Pathologists (CAP): diese US-amerikanische Klassifikation teilt die pathogenen Varianten in 4 Evidenzlevels (engl. tiers) basierend auf den klinischen und prä-klinischen Daten von Studien und unter Berücksichtigung der FDA (Federal Drug Administration) Zulassung ein (7).
- ESMO Scale for Clinical Actionability of Molecular Targets (ESCAT): diese Klassifikation wurde 2018 von der Europäischen Gesellschaft für Medizinische Onkologie (ESMO) eingeführt (8).
- OnkoKB (Precision Oncology Knowledge Base), die erste FDA-anerkannte Tumormutationsdatenbank, die durch das Memorial Sloan Kettering Cancer Center kuratiert wird und die Varianten basierend auf der klinischen Bedeutung und Resistenzstufe einteilt (9).
- NCT-Klassifikation, die für die MTBs innerhalb des Deutschen Krebskonsortiums (DKTK) entwickelt wurde und am Nationalen Centrum für Tumorerkrankungen (NCT) in Heidelberg und anderen deutschen Spitälern angewandt wird (10, 11).
Trotz unterschiedlicher Einstufung sind die Klassifikationssysteme vergleichbar (Tab. 2). Von besonderem Interesse ist der ESCAT Score, weil die Einteilung lediglich auf dem klinischen Evidenzgrad und unabhängig vom internationalen Zulassungsstatus aufgebaut ist. Zum Beispiel, eine PIK3CA-Hotspot-Mutation wird bei Brustkrebs als ESCAT I-A, bei Prostatakrebs als ESCAT II-A und bei nicht-kleinzelligem Lungenkrebs (NSCLC) als ESCAT III-A eingestuft.
Obwohl dieser Score weltweit zunehmend eingesetzt wird, ist seine Anwendung durch das Fehlen eines interaktiven Portals erschwert. Sowohl die Klassifizierung der Varianten als auch die dazugehörige Literatur müssen individuell recherchiert werden, was mit einem erheblichen Zeitaufwand verbunden ist.
In-Label/Off-Label Therapien und klinische Studien
Sowohl die Verfügbarkeit als auch die Kosten müssen bei der therapeutischen Empfehlung im Rahmen des MTB berücksichtigt werden. Bei zielgerichteten Therapien mit nachgewiesenem klinischem Vorteil (z.B. ESCAT I) sind die betroffenen Medikamente im Allgemeinen zugelassen und werden von der Krankenkasse rückvergütet. Für gezielte Therapien mit niedrigerer Evidenzlage (z.B. ab ESCAT II) kann es sein, dass die Substanz nur im «off-label»-Einsatz verfügbar ist. In diesem Fall ist der Patient auf die Gutwilligkeit der Krankenkasse oder der Pharmafirma angewiesen.
Die Teilnahme an klinischen Studien ist grundsätzlich sinnvoll. Auf diese Weise wird die klinische Evidenz erhärtet und der Zugang zu vielversprechenden aber noch nicht erhältlichen Medikamenten kann gewährleistet werden.
Das molekulare Tumorboard ermöglicht unseren Patienten nach Ausschöpfung der Standarttherapien zusätzliche Therapieoptionen. Patient*innen und Therapeut*innen setzen oft viel Hoffnung auf solche Behandlungen, und effektiv scheinen Empfehlungen der MTBs, die klinischen Ergebnisse für Krebspatienten zu verbessern (12–14).
Zusammenfassung
Die klinische Klassifizierung der molekularen Varianten unterstützt die Entscheidungsfindung am molekularen Tumorboard: sie ermöglicht rationale, evidenzbasierte, molekular-pathologisch gestützte Behandlungsempfehlungen abzugeben und bei Vorliegen von mehreren Möglichkeiten die Therapie mit der höchsten Evidenzlage zu bevorzugen.
Copyright bei Aerzteverlag medinfo AG
Luzerner Kantonsspital
Spitalstrasse
6000 Luzern 16
annateresa.allemann@luks.ch
Die Autorin hat keine Interessenkonflikte im Zusammenhang mit diesem Artikel deklariert.
◆ Die therapeutischen Entscheidungen am molekularen Tumorboard beruhen auf der molekularen Charakterisierung und der klinischen Klassifizierung der molekularen Varianten
◆ Für die Auswertung der Pathogenität stehen diverse öffentliche
«In-silico»-Tools zur Verfügung
◆ Die klinische Klassifikation der molekularen Variante ermöglicht,
die therapeutischen Entscheidungen nach der besten Evidenzlage zu steuern
◆ Beim ESCAT-Score basiert die Einteilung ausschliesslich auf dem klinischen Evidenzgrad, unabhängig vom internationalen Zulassungsstatus
1. Meric-Bernstam F, Brusco L, Daniels M, Wathoo C, Bailey AM, Strong L, et al. Incidental germline variants in 1000 advanced cancers on a prospective somatic genomic profiling protocol. Ann Oncol. 2016;27(5):795–800.
2. Lebedeva A, Shaykhutdinova Y, Seriak D, Ignatova E, Rozhavskaya E, Vardhan D, et al. Incidental germline findings during molecular profiling of tumor tissues for precision oncology: molecular survey and methodological obstacles. J Transl Med. 2022;20(1):1–14.
3. Luchini C, Lawlor RT, Milella M, Scarpa A. Molecular Tumor Boards in Clinical Practice. Trends in Cancer. 2020;6(9):738–44.
4. Koopman B, Anthonie, Van Der Wekken J, Arja Ter Elst, Jeroen ; T, Hiltermann N, et al. Relevance and Effectiveness of Molecular Tumor Board Recommendations for Patients With Non-Small-Cell Lung Cancer With Rare or Complex Mutational Profiles. JCO Precis Oncol. 2020. Vol. 4,
5. Richards S, Aziz N, Bale S, Bick D, Das S, Gastier-Foster J, et al. Standards and guidelines for the interpretation of sequence variants: A joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 2015 May 8;17(5):405–24.
6. Pereira R, Oliveira J, Sousa M. Bioinformatics and Computational Tools for Next-Generation Sequencing Analysis in Clinical Genetics. J Clin Med 2020 Jan 1;9(1)
7. Standards and Guidelines for the Interpretation and Reporting of Sequence Variants in Cancer: A Joint Consensus Recommendation of the Association for Molecular Pathology, American Society of Clinical Oncology, and College of American Pathologists. Vol. 19, Journal of Molecular Diagnostics. Elsevier B.V.; 2017. p. 4–23.
8. Mosele F, Remon J, Mateo J, Westphalen CB, Barlesi F, Lolkema MP, et al. Recommendations for the use of next-generation sequencing (NGS) for patients with metastatic cancers: a report from the ESMO Precision Medicine Working Group. Annals of Oncolog 2020. p. 1491–505.
9. With P, Oncokb M, Food US, Comprehensive N, Network C, To R, et al. OncoKB : A Precision Oncology Knowledge Base. JCO Precis Oncol. 2017;(1):1–16
10. Leichsenring J, Horak P, Kreutzfeldt S, Heining C, Christopoulos P, Volckmar AL, et al. Variant classification in precision oncology. Int J Cancer. 2019 Dec 1;145(11):2996–3010.
11. Horak P, Klink B, Heining C, Gröschel S, Hutter B, Fröhlich M, et al. Precision oncology based on omics data: The NCT Heidelberg experience. Int J Cancer. 2017 Sep 1;141(5):877–86.
12. Larson KL, Huang B, Weiss HL, Hull P, Westgate PM, Miller RW, et al. Clinical Outcomes of Molecular Tumor Boards: A Systematic Review. JCO Precis Oncol. 2021;(5):1122–32.
13. Kato S, Kim KH, Lim HJ, Boichard A, Nikanjam M, Weihe E, et al. Real-world data from a molecular tumor board demonstrates improved outcomes with a precision N-of-One strategy. Nat Commun. 2020;11(1):1–9.
14. Gambardella V, Lombardi P, Carbonell-Asins JA, Tarazona N, Cejalvo JM, González-Barrallo I, et al. Molecular profiling of advanced solid tumours. The impact of experimental molecular-matched therapies on cancer patient outcomes in early-phase trials: the MAST study. Br J Cancer. 2021;125(9):1261–9.
info@onco-suisse
- Vol. 12
- Ausgabe 8
- Dezember 2022