- Das molekulare Tumorboard – der zentrale Baustein der Präzisionsonkologie
Tumor profiling ist mittlerweile ein fester Bestandteil der Tumordiagnostik und das molekulare Tumorboard fungiert als essenzieller Knotenpunkt der Präzisionsonkologie zwischen der klinischen Betreuung und den molekularpathologischen Technologien. Im Folgenden gehen wir auf den Ablauf, Chancen und Herausforderungen sowie zukünftige Perspektiven des molekularen Tumorboards ein.
Tumor profiling has become an essential tool in cancer diagnostics and precision oncology and it is the molecular tumorboard that integrates molecular analyses into clinical practice. In this review we address the molecular tumorboard set-up, discuss possibilities and challenges and highlight future perspectives.
Key Words: molecular tumorboard, precision oncology, personalized cancer care
Tumorerkrankungen sind heterogen und weisen eine grosse Vielfalt in ihrer klinischen Präsentation, ihrem biologischen Verhalten und ihrem Therapieansprechen auf – auch innerhalb der gleichen diagnostizierten Entität. Im Rahmen der Präzisionsonkologie strebt man danach, mit Hilfe von genauer Charakterisierung der individuellen Tumorerkrankung ein massgeschneidertes, auf den Patienten abgestimmtes, Behandlungskonzept zu erstellen. Hierbei hat für das Tumor Profiling – die genaue Analyse der genomischen und funktionellen Eigenschaften der Tumorzellen – insbesondere das Next Generation Sequencing (NGS) neue Möglichkeiten einer umfassenden Genanalyse eröffnet. Mittels NGS werden bereits im Rahmen der Erstdiagnose verschiedener Krebserkrankungen sowie auch im weiteren Erkrankungsverlauf molekulare Profile erhoben und tragen hier entscheidend zur Erstellung eines personalisierten Therapieplans bei.
Die Interpretation und Einordnung der molekularen Resultate vor dem individuellen klinischen Hintergrund des Patienten bleibt vielschichtig und bedarf in vielen Fällen einer interdisziplinären Diskussion. Um dieser Herausforderung gerecht zu werden und die komplexen molekularpathologischen Befunde in klinische Handlungsempfehlungen zu übersetzen, wurde an vielen universitären Zentren ein Molekulares Tumorboard (MTB) eingeführt (1-3). An den meisten universitären Zentren der Schweiz wurden erste NGS-Analysen Ende 2014 eingeführt. Die reguläre Testung und das MTB wurden 2015 umgesetzt. MTBs verfolgen das Ziel mittels eines interdisziplinären Vorgehens anhand eines individuellen molekularen Profils potentielle Therapiestrategien zu erarbeiten und somit für ein breites Spektrum von Patienten mit unterschiedlichen Tumorerkrankungen zusätzliche therapierelevante Informationen innerhalb eines nützlichen Zeitrahmens zur Verfügung zu stellen. MTBs können hier zusätzliche Behandlungsempfehlungen nach bereits mehreren erfolgten Behandlungslinien zur Verfügung stellen und das Progressions-freie Überleben für Patienten in dieser Situation verbessern (4,5).
Aktuell erfolgen MTBs über die Institutionen hinweg in einer nicht-standardisierten Form nach hausinternen Richtlinien, sodass der Austausch der Zentren von herausragender Wichtigkeit ist, um ein zukunftsgerichtetes gemeinsames Vorgehen und somit vergleichbare und reproduzierbare Empfehlungen für unsere Patienten sicherzustellen.
Im Folgenden beleuchten wir Schlüsselaspekte der MTB Struktur, aktuelle Chancen und Limitationen und zukünftige Perspektiven anhand der aktuell zur Verfügung stehenden Literatur und anhand unserer Erfahrungen mit dem MTB am Comprehensive Cancer Center Zürich (CCCZ).
Voraussetzungen für die Durchführung eines MTB, Chancen und Limitationen
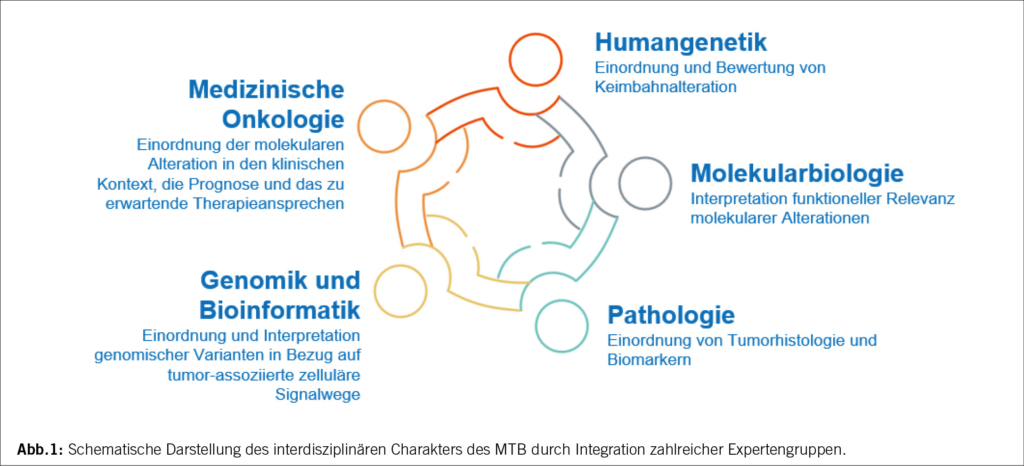
Um eine aufschlussreiche Diskussion der teils komplexen molekularen Befunde zu ermöglichen, ist ein interdisziplinäres Vorgehen unabdingbar. Neben den Medizinischen Onkologen, Pathologen und Molekularpathologen ist der Einblick der Experten der einzelnen molekularpathologischen Technologien sowie der Bioinformatiker und Humangenetiker höchst relevant (Abb. 1). Somit können klinische sowie pathologische und molekularpathologische Besonderheiten eingebracht werden, als auch der ideale Einsatz spezifischer NGS Panels bei der Aufarbeitung individueller Tumorproben und mögliche technische Aspekte, die die Analyse beeinflussen. Weiterhin ist die bioinformatische Annotation der Funktionalität einer gefundenen Alteration ein entscheidender Schritt, um die klinische Interpretation zu ermöglichen. Eine humangenetische Einordnung potentieller Keimbahnalterationen, welche einerseits im Rahmen der vorliegenden Erkrankung treibend oder aber auch als Zufallsbefund erhoben werden können, ist ebenfalls zwingender Bestandteil eines MTBs.
Grundlage für ein MTB am CCCZ ist eine umfassende molekulare Analyse des Tumors. Typischerweise werden am MTB die Resultate aus Tests mit 50-400 Genen besprochen. Im Labor wird die genomische DNA des Tumors entweder aus einer Gewebebiopsie oder dem Blut des Patienten isoliert, die DNA wird modifiziert und auf DNA-Sequenzlesegeräten analysiert. Die Rohdaten werden anschliessend bioinformatisch analysiert, um so pathogene Mutationen des Tumors zu identifizieren. Die pathogenen Mutationen werden in einem übersichtlichen Bericht zusammengefasst, der neben Therapieempfehlungen auch die aktuellen klinischen Studien für bestimmte Mutationen enthält. Im Rahmen des MTBs selbst erfolgt dann eine erneute Interpretation basierend auf dem höchstmöglichen Level der Evidenz – analog internationaler Richtlinien wie beispielsweise der «European Society for Medical Oncology scale for Clinical Actionability» (ESCAT) oder des «Precision Oncology Knowledge Base» (OnkoKB). Die Integration dieser Richtlinien erlaubt Reproduzierbarkeit und internationale Verständlichkeit der Empfehlungen und erleichtert somit den Austausch von Informationen.
Alle Mutationen des Tumorprofiles werden in einer Datenbank erfasst. Hierzu hat das USZ in Anlehnung an das GDC Mutation Annotation Format (MAF) des National Cancer Institutes, USA, ein Datenformat etabliert, um Mutationen zwischen verschiedenen Kliniken auszutauschen (6).
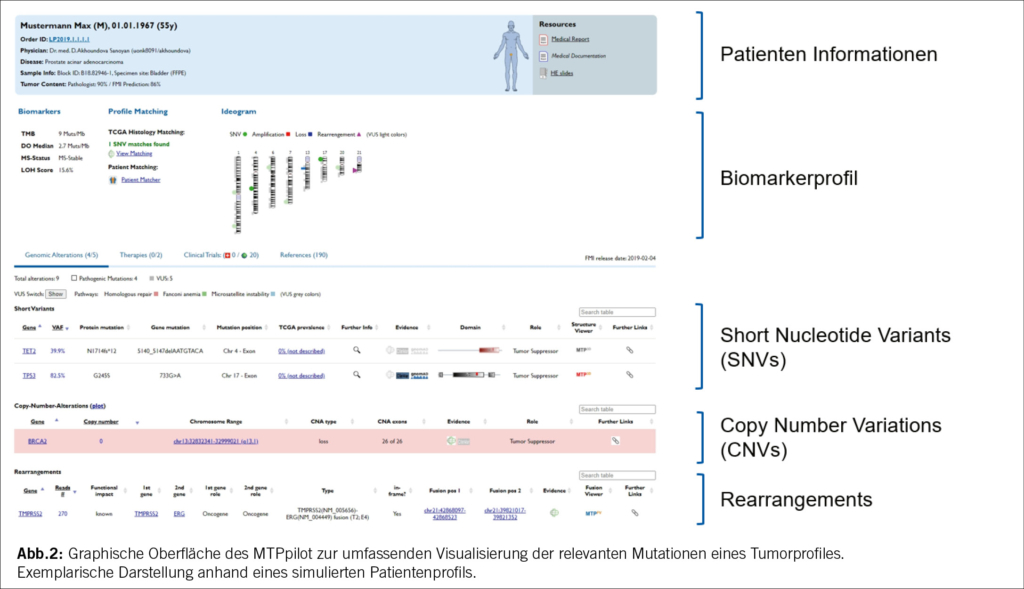
Da für die Besprechung der einzelnen Patienten im MTB jeweils eine begrenzte Zeit zur Verfügung steht, ist es wichtig, alle entscheidungsrelevanten Informationen übersichtlich visualisiert verfügbar zu haben. Für die umfassende und klar strukturierte Darstellung der relevanten Tumormutationen hat das USZ eine Software entwickelt, den MTPpilot (7) (Abb. 2). Der MTPpilot ermöglicht eine schnelle Erfassung aller klinisch relevanten Tumormutationen, um diese im Kontext der Erkrankung zu diskutieren. Weiterhin ist es möglich, über externe Links weitere Informationen zu den genomischen Alterationen in öffentlich zugänglichen Datenbanken abzufragen.
Das Patientengut, das im MTB diskutiert wird, ist aktuell grösstenteils abhängig von der lokalen Vorgehensweise und nicht überregional standardisiert. Üblicherweise werden Tumorpatienten mit Re-biopsien nach Tumorprogress unter bereits erfolgten Standard-of-care Therapien am MTB diskutiert. Weiterhin erfolgen aber auch im Rahmen von Erstdiagnosen bei Erkrankungen mit bekannten onkogenen Treibern sowie bei seltenen Tumortypen molekulare Analysen. Auch longitudinale Testungen mit Frage nach einer erworbenen Resistenzalteration sowie molekulare Vergleiche von metachron aufgetretenen histologisch gegebenenfalls übereinstimmenden Tumoren werden durchgeführt. Tumorspezifische Testalgorithmen werden üblicherweise anhand der bestehenden internationalen Behandlungsrichtlinien durch die lokalen Experten festgelegt und regelmässig überprüft und den neuen Behandlungsoptionen angepasst. Je nach Algorithmus kann ein schrittweises Durchlaufen von Tests erfolgen, die zeitnah die essentiellen Informationen zur Verfügung stellen und kurz darauf auch ein umfassendes molekulares Profil ermöglichen (bspw. Immunhistochemie und NGS hotspot panel bei Erstdiagnose eines nicht-kleinzelligen Lungenkarzinoms mit folgendem umfassenderen NGS Panel im Verlauf).
Teils werden alle, für die am entsprechenden Spital betreuten Patienten, erfolgten molekularpathologischen Analysen im MTB aufgearbeitet, teils nur Fälle mit besonderen Fragestellungen. Dies ist abhängig von den, mit den lokalen Experten erarbeiteten, Richtlinien und natürlich auch den bestehenden Kapazitäten. Am CCCZ wird im Rahmen des wöchentlichen MTBs jeder molekularpathologische Befund von den am Zentrum betreuten Patienten diskutiert und dokumentiert.
Im Rahmen des Berichts des MTBs werden neben der klinischen Geschichte des Patienten, die pathologischen und molekularpathologischen Befunde festgehalten sowie die Diskussion und Empfehlung des MTB Teams. Der Evidenzgrad der Empfehlung wird entweder mittels ESCAT bzw. OnkoKB Level festgehalten oder mittels Quellenangabe.
Die Rolle der MTB Empfehlung bei der Umsetzung von Therapieempfehlungen
Um therapeutische Empfehlungen des MTBs umsetzen zu können, ist insbesondere in «beyond standard of care» und «off label» Situationen eine Kostengutsprache durch die Krankenversicherer notwendig. Hierbei ist die begründete Empfehlung mit Quellenangabe im MTB Bericht hilfreich und kann das Erwirken der Kostenübernahme unterstützen.
Perspektiven für die Zukunft
Strukturierte Datensammlung im MTB
Gemäss einer kürzlich erfolgten Analyse der Daten der OnkoKB Datenbank ist in bis zu 37% aller fortgeschrittenen malignen Tumore mindestens eine potentiell gezielt angehbare onkogene Veränderung nachweisbar (8). Dies legt das Potenzial der Präzisionsonkologie klar dar. Um dieses Potenzial zukünftig optimal für unsere Patienten nutzen zu können ist die Kenntnis über die nachweisbaren Mutationen, die gewählten Therapien und das Therapieansprechen notwendig. Insbesondere vor dem Hintergrund der immensen Heterogenität maligner Tumore, die Genotypen-spezifische klinische Studien verunmöglicht, ist die Etablierung von Dokumentationssystemen anzustreben, um nicht nur fallbasiert im Rahmen des MTBs zu lernen, sondern die Informationen strukturiert sammeln und aufbereiten zu können. Hiermit könnte mittelfristig gegebenenfalls eine zusätzliche Evidenzquelle für molekular-basierte Therapieempfehlungen in Form lokaler, nationaler oder internationaler Daten geschaffen werden. Hierbei sind neben technischen Aspekten (automatisierte Integration diagnostischer Daten, Verwendung international gebräuchlicher Codierungssysteme zur Ermöglichung des Austauschs wie beispielsweise snomed, mcode, FHIR) auch regulatorische Fragen sowie der Datenschutz zu beachten.
Vernetzung
Im Rahmen der Entstehung der MTBs wurden an den universitären Zentren unabhängige Vorgehen definiert und, genau wie für die organspezifischen Tumorboards, unabhängige Instanzen geschaffen. Die Überlegung einer möglichen Vernetzung muss unter der Berücksichtigung der unterschiedlichen Strukturen (bspw. gewähltes Patientengut, angewandte Technologien, unterschiedliche Anbindung an Humangenetik etc.) sowie den steigenden Fallzahlen erfolgen. Vielversprechend scheint insbesondere die punktuelle Vernetzung zur Diskussion spezifischer komplexer Fälle und zum regelmässigen interdisziplinären Abgleich zwischen den Zentren. Dies in Ergänzung zu den lokal erfolgenden regelmässigen MTBs, in welchen der Grossteil der Fälle abgeschlossen würde.
Ein solches Vorgehen wäre per se auch auf internationaler Ebene denkbar und könnte den vorwärtsgewandten und auch hypothesengenerierenden Charakter des MTBs unterstützen und weiterentwickeln. Die Unterschiede zwischen den einzelnen Vorgehen, den verwendeten NGS-Panels und den Zeitpunkten der Analysen sind im internationalen Rahmen allerdings als nochmals bedeutender zu erwarten, könnten aber – gerade aus dieser Diskrepanz heraus – bereichernd sein.
Neue Technologien
Der Nutzen von granulärem multi-omic profiling für individuelle Tumorerkrankungen wird untersucht (9) und neue tumor-profiling Technologien werden entwickelt. So werden beispielsweise die vollständige Exom-Sequenzierung (whole exome sequencing, WES) oder die Sequenzierung des gesamten Genoms (whole genome sequencing, WGS) aktuell zunehmend greifbarer und ein Einsatz in der regulären Tumordiagnostik wird diskutiert (10,11). MTBs sind die institutionellen Werkzeuge, die die Integration dieser Neuerungen ermöglichen, die Präzisionsonkologie somit vorantreiben und die gemeinsame Hypothesengenerierung im Schnittbereich Klinik und Technologie ermöglichen.
Copyright Aerzteverlag medinfo AG
Klinik für Medizinische Onkologie und Hämatologie
Universitätsspital Zürich
Rämistrasse 100
8091 Zürich
Institut für Pathologie und Molekularpathologie
Universitätsspital Zürich
Schmelzbergstrasse 12
8091 Zürich
Klinik für Medizinische Onkologie und Hämatologie
Universitätsspital Zürich
Rämistrasse 100
8091 Zürich
Universität Zürich
Medizinische Fakultät
Pestalozzistrasse 3
8032 Zürich
Die Autoren haben keine Interessenskonflikte im Zusammenhang mit diesem Artikel deklariert.
◆ Das Molekulare Tumorboard ermöglicht die Integration von molekularpathologischen Erkenntnissen in die klinische Patientenbetreuung.
◆ Die Empfehlungen des Molekularen Tumorboards müssen evidenzbasiert sein, hier bieten sich für die Gradierung von (v.a. genetischen) Alterationen die ESCAT und OnkoKB Einstufungen an.
◆ Die Empfehlungen des Molekularen Tumorboards sowie die Umsetzungen und das Therapieansprechen sollte strukturiert erfasst werden (unter Berücksichtigung des Datenschutzes).
◆ Eine nationale und internationale Vernetzung zur Diskussion von besonderen Molekularen Tumorboard-Fällen kann gewinnbringend sein.
◆ Eine Empfehlung des Molekularen Tumorboards mit erläuterter Rationale und Quellenangabe kann ein Kostengutsprachegesuch unterstützen.
1. Tsimberidou AM, Kahle M, Vo HH, Baysal MA et al. Molecular tumour boards – current and future considerations for precision oncology. Nat. Rev. Clin. Oncol. 2023, Dec 20:843-863.
2. Tamborero D, Dienstmann R, Rachid MH et al. The Molecular Tumor Board Portal supports clinical decisions and automated reporting for precision oncology. Nat Cancer. 2022 Feb;3(2):251-261.
3. Mateo J, Chakravarty D, Dienstmann R et al. A framework to rank genomic alterations as targets for cancer precision medicine: the ESMO Scale for Clinical Actionability of molecular Targets (ESCAT). Ann Oncol. 2018 Sep 1;29(9):1895-1902.
4. Hoefflin R, Lazarou A, Hess ME et al. Transitioning the Molecular Tumor Board from Proof of Concept to Clinical Routine: A German Single-Center Analysis. Cancers (Basel). 2021 Mar 8;13(5):1151.
5. Kato S, Kim KH, Lim HJ et al. Real-world data from a molecular tumor board demonstrates improved outcomes with a precision N-of-One strategy. Nat Commun. 2020 Oct 2;11(1):4965.
6. National Cancer Institute, https://docs.gdc.cancer.gov/Data/File_Formats/MAF_Format/
7. Kahraman A, Arnold F, Hanimann J et al. MTPpilot: An Interactive Software for Visualization of Next-Generation Sequencing Results in Molecular Tumor Boards. JCO Clin Cancer Inform 6:e2200032 (2022).
8. Zehir A, Benayed R, Shah RH et al. Mutational landscape of metastatic cancer revealed from prospective clinical sequencing of 10,000 patients. Nat Med. 2017 Jun;23(6):703-713.
9. Irmisch A, Bonilla X, Chevrier S et al. The Tumor Profiler Study: integrated, multi-omic, functional tumor profiling for clinical decision support. Cancer Cell. 2021 Mar 8;39(3):288-293.
10. Simons M, Van De Ven M, Coupé V et al. Early technology assessment of using whole genome sequencing in personalized oncology. Expert Rev Pharmacoecon Outcomes Res. 2021 Jun;21(3):343-351.
11. Samsom KG, Bosch LJW, Schipper LJ, Schout D et al. Optimized whole-genome sequencing workflow for tumor diagnostics in routine pathology practice. Nat Protoc. 2024 Mar;19(3):700-726.
info@onco-suisse
- Vol. 14
- Ausgabe 4-5
- August 2024