- Tumormutationsprofilierung
Moderne Sequenziertechnologien (Next Generation Sequencing, NGS) ermöglichen eine umfassende genetische Analyse von Tumorzellen (Tumormutationsprofilierung). Veränderungen im Tumorzellgenom werden als Biomarker in der Präzisionsonkologie verwendet. Der nachfolgende Beitrag beschreibt Anwendungen der Tumormutationsprofilierung in der klinischen Diagnostik von onkologischen Erkrankungen.
Les technologies modernes de séquençage (Next Generation Sequencing, NGS) permettent une analyse génétique complète des cellules tumorales (profil de mutation tumorale). Les modifications du génome des cellules tumorales sont utilisées comme biomarqueurs en oncologie de précision. L’article suivant décrit les applications du profilage des mutations tumorales dans le diagnostic clinique des maladies oncologiques.
The tumor mutation profile is the totality of aberrations in the genome of a tumor. Recently, the pan-cancer atlas was published, which describes a comprehensive characterization of 33 tumor entities at the DNA and RNA levels (1). Acquired somatic mutations are key mechanisms in the development of solid tumors and haemato-oncological diseases (2). In addition to simple sequence variants, structural and numerical gene changes are differentiated.
In clinical tumor diagnostics, the focus is on the analysis of tumor-associated genes in which mutations with diagnostic, prognostic and / or predictive significance can occur (3).
Methodological aspects
For mutational profiling of tumors, high-throughput sequencing (NGS) is used, which allows parallel analysis of numerous target regions at the DNA and RNA levels (Figure 1) (4). In clinical applications, especially panels of up to several hundred tumor-associated genes are being analyzed. Mutation detection is performed by comparing the sequencing results with the corresponding sections of a human reference genome (hg19, GRCh38). For the description of sequence variants, a standardized nomenclature of HGVS is available (5). By means of suitable bioinformatics instruments NGS can also be used to detect numerical and structural changes.
Mutational profiling can be performed on paraffin-embedded tissue samples, cytological specimens, bone marrow aspirates, and tumor cells in blood samples (especially leukemias / lymphomas). Before DNA / RNA extraction, sufficient tumor cell content of the sample material must be ensured or a targeted tumor cell enrichment performed.
Assessment of the clinical relevance of a mutation
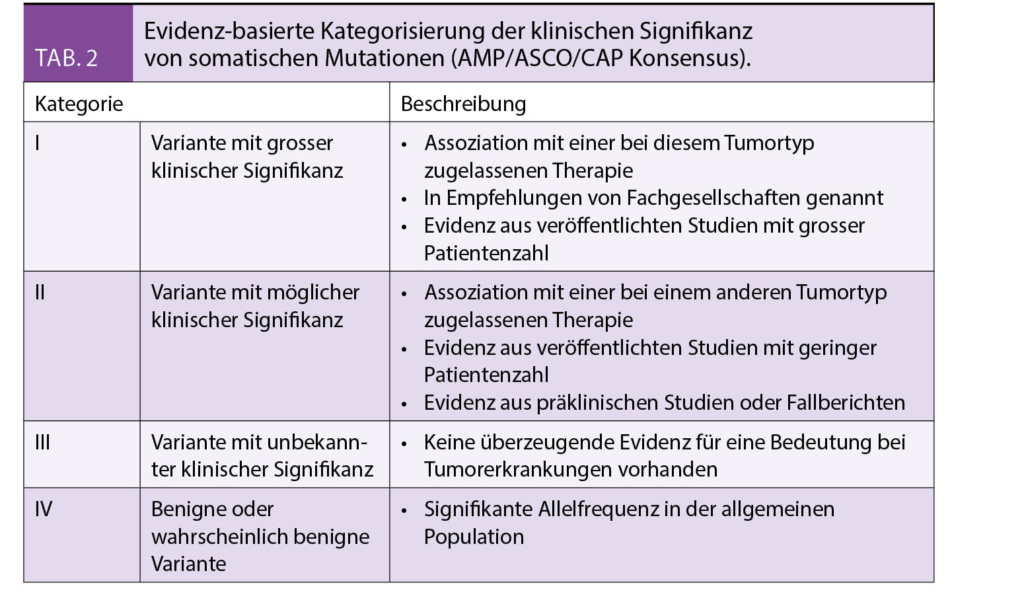
Die Pathogenität einer somatischen Mutation kann analog zu Keimbahnveränderungen klassifiziert werden (6). Bedeutsamer ist die Beurteilung der Actionability einer Mutation. Eine Mutation wird als actionable bezeichnet, falls sie ein Ansprechen oder eine Resistenz auf eine zugelassene oder experimentelle Therapie voraussagt bzw. als diagnostischer und/oder prognostischer Marker verwendet werden kann (Tab. 1) (7). Informationen zur Actionability einer Mutation finden sich in öffentlich zugänglichen Online Datenbanken (MyCancerGenome.org, PersonalizedCancerTherapy.org, Oncokb.org) (8). Kürzlich wurde im Rahmen eines AMP/ASCO/CAP Konsensus eine Evidenz-basierte Kategorisierung von somatischen Varianten/Mutationen in Hinblick auf ihre diagnostische, prognostische und/oder therapeutische Bedeutung vorgeschlagen (Tab. 2) (9).
Tumorklassifikation und Risikostratifizierung
Aktuelle Klassifikationssysteme verwenden vor allem phänotypische Merkmale zur Tumoreinteilung. Bei der Definition und Namensgebung von Tumorentitäten werden jedoch zunehmend auch genotypische Merkmale der Tumorzellen berücksichtigt. Beispiele finden sich in den aktuellen WHO Klassifikationen der ZNS Tumore, Leukämien/Lymphome und Weichgewebstumore. Die Mutationsprofilierung bietet sich an, wenn ein Tumor gleichzeitig auf Veränderungen in mehreren Genen untersucht werden muss, um eine abschliessende diagnostische und/oder prognostische Einordnung zur erreichen.
Identifikation von Patienten für etablierte zielgerichtete Therapien
Bei Tumortypen, bei denen mehrere klinisch relevante Mutationen vorliegen können, werden durch eine parallele Testung auf verschiedene genomische Aberrationen Effizienzgewinne und eine kürzere Bearbeitungszeit im Vergleich zu einer sequenziellen Einzelgentestung erzielt. So lassen sich histologisch gleichartige nicht-kleinzellige Lungenkarzinome (NSCLC) durch den Nachweis von charakteristischen Treibermutationen in molekulare Subtypen unterteilen, für die spezifische zielgerichtete Therapien verfügbar sind (10, 11). Durch eine Mutationsprofilierung können neben Veränderungen in den EGFR und ALK Genen auch seltene Treibermutationen (BRAF, HER2, MET, RET, NTRK1/2/3) detektiert werden.
Identifikation von Patienten für experimentelle Therapien
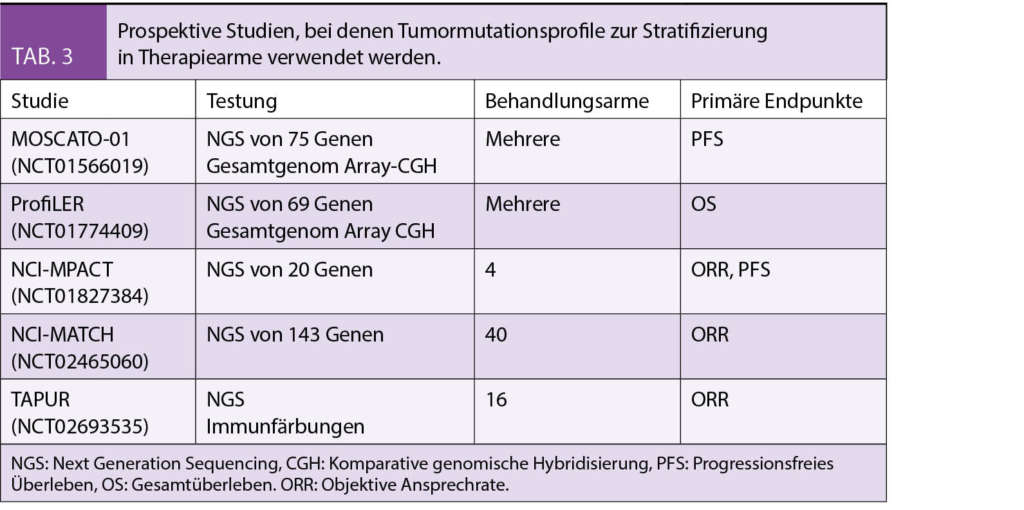
Bei onkologischen Therapiestudien mit «Baske» oder «Umbrella» Design erfolgt die Stratifizierung in die verschiedenen experimentellen Behandlungsarme anhand genotypischer Tumormerkmale (12). Das Mutationsscreening mittels zielgerichteter NGS ist ein effizientes Verfahren, Patienten für einen Studieneinschluss zu identifizieren. Die Machbarkeit und der klinische Nutzen einer systematischen Anwendung wird gegenwärtig in mehreren prospektiven Studien (MOSCATO-01, NCI-MPACT, NCI-MATCH, ProfiLER, TAPUR, etc.) unter Verwendung von klinisch relevanten Endpunkten wie Ansprechrate und Überlebenszeit untersucht (Tab. 3).
Identifikation von Patienten für eine Therapie mit Immuncheckpunkt-Inhibitoren
Während bei zielgerichteten Therapien der Nachweis bestimmter Mutationen von Bedeutung ist, so scheint bei Immuncheckpunkt-Inhibitoren die sogenannte Mutationslast eines Tumors (TML) mit dem Therapieansprechen assoziiert zu sein (13). Während die TML initial mittels Gesamt-Exom-Sequenzierung (WES) untersucht wurde, deuten aktuelle Studienergebnisse bei NSCLC darauf hin, dass die TML auch durch Sequenzierung eines kleineren genomischen Fussabdrucks (mindestens 1 Megabase) ermittelt werden kann (14, 15).
Erbliche Tumorprädisposition
Die Tumormutationsprofilierung zielt auf die Identifikation von somatischen Genaberrationen. Ohne vergleichende Untersuchung von Nichttumorgewebe (oder Blutzellen) des gleichen Individuums kann jedoch bei positivem Mutationsnachweis nicht sicher zwischen einer bei der Tumorentstehung erworbenen Mutation und einer Keimbahnmutation unterschieden werden. Hinweise auf das Vorliegen einer Keimbahnmutation ergeben sich aus der Allelfrequenz einer Variante unter Berücksichtigung des Tumorzellgehalts des Probenmaterials. Klinisch relevant sind vor allem Mutationen in den Genen, die mit einer erblichen Tumorprädisposition assoziiert sind (16).
Regulatorische Aspekte
Somatische Mutationsanalysen fallen zukünftig in den Geltungsbereich des revidierten Gesetzes über genetische Untersuchungen beim Menschen (GUMG). Daraus ergeben sich neue regulatorischen Anforderungen auch für die molekulare Tumordiagnostik (Patientenaufklärung/-einwilligung, Vermeidung von Überschussinformation, Informationspflicht bei inzidentellen Ergebnissen, etc.).
Herausforderungen
Die Tumormutationsprofilierung mittels NGS ist mit grossen infrastrukturellen, personellen und organisatorischen Anforderungen verbunden, um kurze Bearbeitungszeiten und eine gleichbleibend hochstehende Ergebnisqualität zu erreichen. Besondere Herausforderungen ergeben sich bezüglich der bioinformatischen Datenanalyse und der Datenspeicherung.
Die Beurteilung der Actionability von Tumorzellmutationen bleibt trotz der Verfügbarkeit von öffentlich zugänglichen Datenbanken und Online-Werkzeugen schwierig. Der Inhalt von Datenbanken entspricht bestenfalls dem Wissensstand zum Zeitpunkt der Abfrage. Mit der Anzahl der untersuchten Genregionen nimmt auch die Zahl der Sequenzvarianten zu, deren funktionelle Signifikanz unbekannt ist (VUS) und deren klinische Validierung als diagnostischer, prognostischer oder prädiktiver Biomarker bislang fehlt.
Schlussfolgerungen und Ausblick
Die Mutationsprofilierung hat sich als nützliches Werkzeug der molekularen Tumordiagnostik etabliert. Hauptanwendung ist die Analyse von Biomarkern für die Diagnostik (Klassifikation), Prognoseabschätzung und Vorhersage eines Therapieansprechens bei onkologischen Erkrankungen. In der Schweiz wird die Tumormutationsprofilierung von mehreren Pathologie-Instituten angeboten. Die angebotenen Genpanel unterscheiden sich hinsichtlich Grösse und Zusammensetzung.
Trotz aller Fortschritte bei der Standardisierung (Nomenklatur, Berichtswesen, etc.) bleibt die direkte interdisziplinäre Interaktion zwischen diagnostisch und therapeutisch ausgerichteten Fachspezialisten unabdingbar, um die klinische Signifikanz molekularer Testergebnisse im Kontext einer individuellen Patientensituation zu bewerten und Therapieempfehlungen im Einzelfall zu entwickeln. Mögliche zukünftige Entwicklungen sind die Mutationsprofilierung zirkulierender Tumor-DNA (ctDNA) und die Einführung der Gesamt-Exom-Sequenzierung (WES) in die klinische Tumordiagnostik.
Institut für Pathologie
Kantonsspital St. Gallen
9007 St. Gallen
wolfram.jochum@kssg.ch
Der Autor gibt an, dass für diese Publikation kein Interessenskonflikt vorliegt.
- Das Mutationsprofil eines Tumors beschreibt die Gesamtheit der genetischen Veränderungen in den Tumorzellen.
- Zum Nachweis von Mutationen in tumorassoziierten Genen wird im klinischen Kontext vor allem die zielgerichtete Hochdurchsatz-Sequenzierung (NGS) eingesetzt.
- Hauptanwendungen der Tumormutationsprofilierung sind die Tumorklassifikation, die Risikostratifizierung/Prognoseabschätzung und die Vorhersage eines Ansprechens auf zielgerichtete Therapien und Immuncheckpunkt-Inhibition.
Messages à retenir
- Le profil de mutation d’une tumeur décrit tous les changements génétiques dans les cellules tumorales.
- Dans le contexte clinique, le séquençage ciblé à haut débit (NGS) est principalement utilisé pour détecter les mutations des gènes associés aux tumeurs.
- Les principales applications du profilage des mutations tumorales sont la classification des tumeurs, la stratification des risques/estimation des prévisions et la prédiction de la réponse aux traitements ciblés et l’immunochimie.
1. Hoadley KA, Yau C, Hinoue T, Wolf DM, Lazar AJ, Drill E, Shen R, et al. Cell-of-Origin Patterns Dominate the Molecular Classification of 10,000 Tumors from 33 Types of Cancer. Cell 2018;173:291-304 e296.
2. Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell 2011;144:646-674.
3. Hyman DM, Taylor BS, Baselga J. Implementing Genome-Driven Oncology. Cell 2017;168:584-599.
4. Reuter JA, Spacek DV, Snyder MP. High-throughput sequencing technologies. Mol Cell 2015;58:586-597.
5. den Dunnen JT, Dalgleish R, Maglott DR, Hart RK, Greenblatt MS, McGowan-Jordan J, Roux AF, et al. HGVS Recommendations for the Description of Sequence Variants: 2016 Update. Hum Mutat 2016;37:564-569.
6. Richards S, Aziz N, Bale S, Bick D, Das S, Gastier-Foster J, Grody WW, et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med 2015;17:405-424.
7. Meric-Bernstam F, Johnson A, Holla V, Bailey AM, Brusco L, Chen K, Routbort M, et al. A decision support framework for genomically informed investigational cancer therapy. J Natl Cancer Inst 2015;107.
8. Prawira A, Pugh TJ, Stockley TL, Siu LL. Data resources for the identification and interpretation of actionable mutations by clinicians. Ann Oncol 2017;28:946-957.
9. Li MM, Datto M, Duncavage EJ, Kulkarni S, Lindeman NI, Roy S, Tsimberidou AM, et al. Standards and Guidelines for the Interpretation and Reporting of Sequence Variants in Cancer: A Joint Consensus Recommendation of the Association for Molecular Pathology, American Society of Clinical Oncology, and College of American Pathologists. J Mol Diagn 2017;19:4-23.
10. Hanna N, Johnson D, Temin S, Baker S, Jr., Brahmer J, Ellis PM, Giaccone G, et al. Systemic Therapy for Stage IV Non-Small-Cell Lung Cancer: American Society of Clinical Oncology Clinical Practice Guideline Update. J Clin Oncol 2017;35:3484-3515.
11. Jordan EJ, Kim HR, Arcila ME, Barron D, Chakravarty D, Gao J, Chang MT, et al. Prospective Comprehensive Molecular Characterization of Lung Adenocarcinomas for Efficient Patient Matching to Approved and Emerging Therapies. Cancer Discov 2017;7:596-609.
12. Senft D, Leiserson MDM, Ruppin E, Ronai ZA. Precision Oncology: The Road Ahead. Trends Mol Med 2017;23:874-898.
13. Gibney GT, Weiner LM, Atkins MB. Predictive biomarkers for checkpoint inhibitor-based immunotherapy. Lancet Oncol 2016;17:e542-e551.
14. Rizvi H, Sanchez-Vega F, La K, Chatila W, Jonsson P, Halpenny D, Plodkowski A, et al. Molecular Determinants of Response to Anti-Programmed Cell Death (PD)-1 and Anti-Programmed Death-Ligand 1 (PD-L1) Blockade in Patients With Non-Small-Cell Lung Cancer Profiled With Targeted Next-Generation Sequencing. J Clin Oncol 2018;36:633-641.
15. Hellmann MD, Ciuleanu TE, Pluzanski A, Lee JS, Otterson GA, Audigier-Valette C, Minenza E, et al. Nivolumab plus Ipilimumab in Lung Cancer with a High Tumor Mutational Burden. N Engl J Med 2018;378:2093-2104.
16. Kalia SS, Adelman K, Bale SJ, Chung WK, Eng C, Evans JP, Herman GE, et al. Recommendations for reporting findings in clinical exome and genome sequencing, 2016 update (ACMG SF v2.0): a policy statement from the American College of Medical Genetics and Genomics. Genet Med 2017; 19: 249-255.
info@onco-suisse
- Vol. 9
- Ausgabe 3
- Juni 2019