- Wegweiser durch die Präzisionsmedizin?
Tumormarker – ihre Zahl steigt täglich, zumindest gemessen an den Publikationszahlen. In die klinische Routine haben jedoch nur die wenigsten Einzug gehalten. Während die Präzisionsmedizin auf neue akkurate Marker dringend angewiesen ist, bleiben die altbekannten statistischen Masszahlen Sensitivität, Spezifität, Prävalenz und positiv prädiktiver Wert oft unüberwindliche Hindernisse auf dem Weg der Translation. Hoffnung gibt es jedoch bei neuen Anwendungsgebieten, z.B. in der Vorhersage des Therapieansprechens oder der Prognose und Prävention.
Les marqueurs tumoraux – leur nombre augmente chaque jour, au moins mesuré par les chiffres de publication. Dans la routine clinique, cependant, le le plus petit d’entre eux. Alors que la médecine de précision a besoin de toute urgence de nouveaux marqueurs précis, les nombres bien connus, la sensibilité, la spécificité, la prévalence et la valeur prédictive positive souvent sont des obstacles insurmontables sur le chemin de la translation. Un espoir reste dans les nouveaux domaines d’application, par exemple dans la prédiction de l’évolution de la réponse thérapeutique ou le pronostic et la prévention.
Der neueste «Hype» in der Medizin, die «Präzisionsmedizin», setzt sich zum Ziel, jedem Patienten eine auf ihn abgestimmte Therapie anbieten zu können – dies bedarf nicht nur massgeschneiderter Therapien, sondern auch einer spezifischen Diagnostik – im Fall der Onkologie einer ganzen Batterie neuer «Tumormarker»(1, 2).
Tumormarker sind mess- oder berechenbare Entitäten, die sensitiv, spezifisch und ohne Bias die Prädisposition, das Auftreten, die Progression, das Therapieansprechen oder die Abwesenheit eines Tumors oder seines Surrogates bezeichnen (3–5). Unter eine solche Definition fallen neben den klassischen klinisch-chemischen Einzelanalysen auch Ratios von freien und totalen Markern (z.B. totales und freies PSA) (6), Anstiegsgeschwindigkeiten (z.B. beim PSA (7)), Scores (8), komplexe Metabolitmuster (9) und Algorithmen (10). Aufgrund der Vielzahl der Anwendungsgebiete greift jedoch der Begriff des «Tumormarkers» zu kurz, denn ein Marker, der sensitiv die Präsenz einer Malignität anzeigt, ist für eine Prognoseabschätzung möglicherweise ungeeignet oder ein genetischer Marker, der mit grosser Sicherheit die Empfindlichkeit bzw. Resistenz eines Tumors gegenüber einer spezifischen Behandlung voraussagt, ist unter Umständen für die Verlaufsbeobachtung unbrauchbar.
Prädispositionsmarker
In den letzten Jahren wurde eine grosse Menge genetischer Risikofaktoren aufgedeckt, die als gemeinsame oder spezifische Risikofaktoren für die Entstehung von malignen Tumoren in Frage kommen (11). Zu den bekanntesten zählen BRCA-Mutationen für das Mammakarzinom (12, 13) oder hereditäre Kolonkarzinome (14). Während die aktuellen Ansätze des Whole-Genome-Sequencings (WGS) immer neue Mutationen und Varianten ans Licht bringen, bleibt die klinische Wertigkeit solcher Befunde häufig unklar. Daraus entstehen Herausforderungen für die moderne Medizin: zum einen an die Aufarbeitung und Übermittlung (15, 16) von Daten mit potenziell prädisponierender Information, zum andern an den Umgang mit aufgeklärten Patienten (vgl. «patients like me» ), aber auch an die Regularien für die Befundmitteilung, wie z.B. ab welchem Grad an Verlässlichkeit der Patient informiert werden sollte. Für klinische Studien (17) und nationale Verbundprojekte, wie z.B. im Rahmen des Swiss Personalized Health Networks (SPHN) sind diese Fragen essentiell.
Da sich insbesondere auf dem Gebiet der genetisch bedingten Tumoren die Datenlage extrem schnell ändert, ist es empfehlenswert, sich mit Hilfe der Literatur (z.B. aktuelle Guidelines(18, 19)), molekularen Tumorboards (20), in Tumorkonferenzen unter Beteiligung molekularer Pathologen und Genetiker und mit modernen Werkzeugen des Risikoassessments (21)«up-to-date» zu halten.
Marker für die Früherkennung
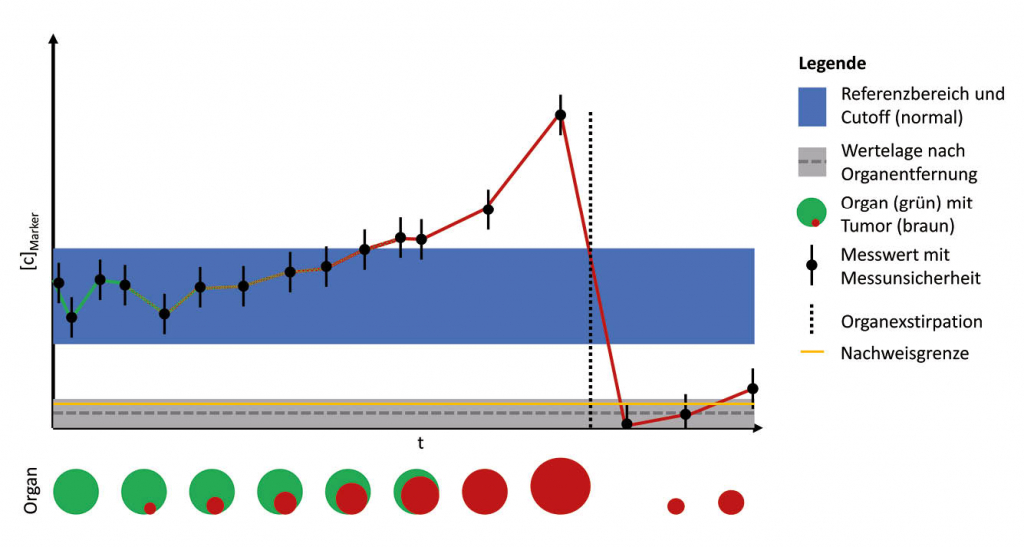
Die Früherkennung von Tumorerkrankungen ist von herausragender Bedeutung, da viele Tumoren, wenn sie denn rechtzeitig erkannt werden, durch chirurgische und häufig auch konventionelle Verfahren effektiv behandelt und ggf. sogar geheilt werden können (22). Daher steht die Nutzung von Tumormarkern zur Früherkennung von Karzinomen im Interesse der aktuellen Forschung. Eine inzwischen unüberschaubare Anzahl an Publikationen «entdeckt» immer neue Frühmarker und nutzt dazu hochkomplexe und für jedweden Bias anfällige «-omics»-Technologien (5, 23). Häufig wird dabei aber vergessen, dass sich Tumoren, und damit auch die meisten Tumormarker, von ursprünglich gesundem Gewebe ableiten – deswegen haben insbesondere die «klassischen» Tumormarker Referenzbereichsgrenzen, welche die Variabilität der Marker in der Population abbilden. Hori und Gambhir (24) konnten mit Hilfe einer mathematischen Modellierung nachweisen, dass z.B. ein Ovarialkarzinom einen Durchmesser von ca. 2.5cm erreichen muss, bevor CA125 als Tumormarker den Referenzbereich überschreitet – dies würde einer «lead-time» von 10 bis 12 Jahren entsprechen. Von Früherkennung kann hier keine Rede sein (vgl. Abbildung 1). Generell haben die Entwicklungen der letzten 20 Jahre eher enttäuschende Ergebnisse geliefert (25, 26). Einen Ausweg könnten neue labormedizinische Technologien bieten, so z.B. die sog. «circulating tumor cells» (CTCs), «circulating tumor DNA” (ctDNA), «cell free RNA» (cfRNA), «tumor-educated platelets» (TEPs) und Exosomen im Rahmen der sog. «liquid biopsy». Weil Tumoren mit spezifischen Mutationen einhergehen, die im gesunden Organ nicht vorkommen, können Tumorgenom-Sequenzen aus wenigen Zellen, Thrombozyten oder Exosomen im Blut amplifiziert und so ausgeschwemmte Tumorzellen im peripheren Blut nachgewiesen z. B. werden (27). Aktuell stellt die geringe Konzentration der genetischen Marker im Blut noch eine technische Herausforderung für die Analytik dar (28).
Screening
Noch kritischer als für die Früherkennung sind die Anforderungen für Tumormarker, wenn sie für ein populationsbasiertes Screening eingesetzt werden sollen. Die in der generellen Bevölkerung niedrige Prävalenz von Tumorerkrankungen führt auch bei ausserordentlich hoher Spezifität zu einer kritischen Anzahl falsch positiver Befunde, die in der Regel eine belastende Folgediagnostik nach sich zieht. Eine gute Masszahl zur diagnostischen Einschätzung von Tumormarkern (29) bietet daher der positiv prädiktive Wert (PPV, «positive predictive value»), der angibt, mit welcher Wahrscheinlichkeit ein positives Testergebnis auch indikativ für die Erkrankung ist. Für die Beurteilung klinischer Konsequenzen der Anwendung eines Tumormarkertests bietet sich z.B. die Decision-Curve-Analysis (DCA) an (30). Während die Verwendung von Tumormarkern für Screeningzwecke in der älteren Literatur mehr als zurückhaltend betrachtet wird (25, 31), zeigen neuere Arbeiten, dass sich mit Hilfe von rechnergestützten Simulationen, ausgehend von initialen Basiswerten und ihrer Variabilität, die Tumordetektionszeiten verkürzen lassen, wenn eine grössere falsch-positiv-Rate (FPR) in Kauf genommen wird (32, 33). Dies lässt die Vermutung zu, dass aufgrund der hohen inter-individuellen Variabilität von Tumormarker-Konzentrationen ein in gesundem Zustand gemessener, individueller Ausgangswert, auch in Anbetracht der FPR für die Beurteilung eines späteren Anstiegs hilfreich sein könnte. An dieser Stelle sei auch darauf hingewiesen, dass eine Vielzahl an präanalytischen Einflussgrössen und Störfaktoren die gemessenen Markerkonzentrationen beeinflussen kann (25) und dass sich für dieselbe Probe je nach Hersteller des Testkits die Werte erheblich unterscheiden können, auch wenn das Verfahren selbst prinzipiell vergleichbar ist (34).
Diagnostische Marker
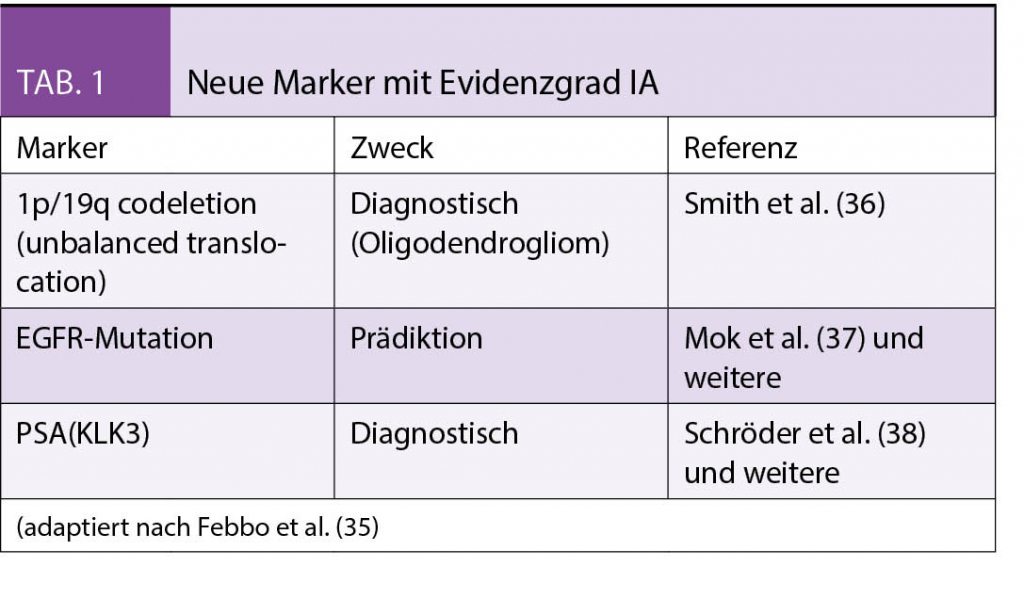
Neben den seit Jahrzehnten bewährten Tumormarkern (25) haben trotz ihrer bekannten Limitationen (31) nur wenige neue Marker den Sprung in die Routinediagnostik geschafft (35): Belastbare, evidenzbasierte Daten (Evidenzstufe I [Markerstudie mit ausreichend Power bzw. Meta-Analyse] der Kategorie A [prospektive Evaluationsstudie]) liegen für neue Marker kaum vor (siehe Tabelle 1).
Für eine Übersicht über die aktuell in der Routine verwendeten Tumormarker sei auf die Übersichtsliteratur verwiesen (39–42).
Companion Diagnostics
Als Companion Diagnostics werden in vitro-Diagnostika bezeichnet, die Informationen für die sichere und effektive Nutzung eines korrespondierenden therapeutischen Produkts bereitstellen (43): Häufig werden Companion Diagnostics zusammen mit einem Therapeutikum entwickelt und hergestellt. Während der präklinischen Studienphasen werden Biomarker evaluiert, die das Therapieansprechen im individuellen Patienten vorhersagen können und selbige dann in späteren klinischen Phasen analytisch und klinisch evaluiert. Ihr Hauptanwendungsgebiet finden Companion Diagnostics derzeit in der Onkologie, prinzipiell sind sie aber für das gesamte Spektrum der personalisierten Medizin interessant (44). Durch ihren konsequenten Einsatz können oft sehr teure, unnötige Therapiekosten eingespart werden (45), wobei sich die Aufwandsbetrachtung Therapien dabei jeweils auf die Gesamtkosten beziehen sollte. In Zukunft dürfte sich die Zahl der als Companion Diagnostics bezeichneten Marker im Zuge der Personalisierung der Tumortherapie rapide erhöhen, was im Gegenzug eine Validierung mit ausreichend grossen Kohorten zur Herausforderung werden lässt. Für die korrekte Auswahl, Messung und Interpretation gewinnt eine patientenzentrierte, interdisziplinäre Zusammenarbeit von Klinikern sowie von Pathologen und Labormedizinern immer grössere Bedeutung – ein Umstand, dem z.B. in Bern mit der Neugründung eines integrativen Clinical Genomics Lab (CGL) Rechnung getragen wird.
Prognosemarker
Neben der rein diagnostischen Funktion kommt einer Reihe von Tumormarkern auch eine prognostische Aussagekraft zu, so z.B. dem CEA, CA125, S100A4 und Mesothelin beim Pankreaskarzinom (46, 47), ein Panel von 26 histologischen Markern bei Lebermetastasen einem kolorektaler Karzinome (48), oder zirkulärer RNA beim hepatozellulären Karzinom (49) bzw. epigenetische Marker bei unterschiedlichen Tumoren(50), um nur einige wenige epigenetischen Markern Beispiele zu nennen. Während der Nutzen für den Patienten, der bei jeder Diagnostik im Vordergrund stehen sollte, bei einer rein prognostischen Wertigkeit fraglich bleibt, käme einer präventiven Therapie unter regelmässiger Überwachung durch geeignete Marker, die derzeit noch weitgehend fehlen, eine grosse Bedeutung zu (51).
Choosing Wisely
Wie geht es weiter mit den Tumormarkern? Angesichts der Fülle jedes Jahr publizierter neuer Marker und der Schwierigkeit, die Validität von Markerstudien im Einzelfall zu erfassen, kommt Initiativen wie «Choosing Wisely» (19) eine immer grössere Bedeutung zu. Eine Gruppe Schweizer Autoren hat kürzlich im Journal der European Society for Medical Oncology eine Übersichtsarbeit (52) verfasst, die sich mit Strategien befasst, wie unangemessene Diagnostik und Behandlung vermieden und die Fachdisziplinen in einen produktiven Dialog gebracht werden können. Als besondere Hemmnisse identifizieren sie Voreingenommenheiten, die in einem hierarchisch organisierten Medizinsystem wohl erst mit ihren Vertretern Schritt für Schritt weniger werden, daneben aber auch eine «defensive», innovationsfeindliche Grundhaltung in der Medizin und Interessenkonflikte. Als mögliche Lösungsansätze sehen sie nicht nur eine Ausbildung künftiger Ärzte in medizinischer Entscheidungsfindung, sondern auch in der Einführung von elektronischen Werkzeugen, die die Adhärenz an Guidelines zu einem gewissen Grad automatisieren und damit so vereinfachen, dass davon abzuweichen schwerer fällt, als sich daran zu halten. Solche automatisierten Diagnostik- und Therapieempfehlungen werden in Zukunft ein unverzichtbarer Wegweiser durch das immer undurchdringlichere Dickicht aus neuen Markern und Therapien und dazu beitragen, den Patienten eine passende, auf sie personalisierte Medizin zu ermöglichen.
Universitätsinstitut für Klinische Chemie und Insel Data Science Center
Inselspital – Universitätsspital Bern und Universität Bern
3010 Bern
Universitätsinstitut für Klinische Chemie
Inselspital – Universitätsspital Bern und Universität Bern
3010 Bern
Die Autoren geben an, dass für diese Publikation kein Interessenkonflikt vorliegt.
- Bei einer sehr niedrigen Erkrankungsprävalenz wird für Screeninganwendungen die falsch-positiv– Rate auch bei guter Spezifität eines Tumormarkers zum Problem.
- Die Aussagekraft eines Tumormarkers hängt wesentlich von seinem Anwendungszweck (z.B. Verlaufsbeurteilung, Screening) ab.
- Die grosse Mehrheit der alljährlich publizierten “Tumormarker” erreicht keine Evidenzgrade, die eine schnelle Einführung in die klinische Routine erlauben.
Messages à retenir
- Avec une très faible prévalence de la maladie, le taux de faux positifs devient un problème pour le dépistage des applications même avec une bonne spécificité d’un marqueur tumoral.
- L’importance d’un marqueur tumoral dépend essentiellement de son utilisation prévue (p. ex. évaluation des progrès, dépistage).
- La grande majorité des “marqueurs tumoraux” publiés chaque année n’atteignent aucun niveau de preuve permettant une introduction rapide dans la routine clinique.
1. Shrager J, Tenenbaum JM. Rapid learning for precision oncology. Nat Rev Clin Oncol. 2014;11:nrclinonc.2013.244.
2. Ahlquist DA. Universal cancer screening: revolutionary, rational, and realizable. Npj Precis Oncol. 2018;2:23.
3. Diamandis EP. Cancer Biomarkers: Can We Turn Recent Failures into Success? Jnci J National Cancer Inst. 2010;102:1462–7.
4. Leichtle A, Dufour J-F, Fiedler GM. Potentials and pitfalls of clinical peptidomics and metabolomics. Swiss Med Wkly. 2013;143:w13801.
5. Leichtle A. Biomarker – vom Sein und Wesen. Laboratoriumsmedizin. 2015;39:97–101.
6. Münzker J, Hofer D, Trummer C, Ulbing M, Harger A, Pieber T, et al. Testosterone to Dihydrotestosterone Ratio as a New Biomarker for an Adverse Metabolic Phenotype in the Polycystic Ovary Syndrome. J Clin Endocrinol Metabolism. 2015;100:653 660.
7. Antonarakis ES, Zahurak ML, Lin J, Keizman D, Carducci MA, Eisenberger MA. Changes in PSA kinetics predict metastasis- free survival in men with PSA-recurrent prostate cancer treated with nonhormonal agents: combined analysis of 4 phase II trials. Cancer. 2012;118:1533 1542.
8. Hemelrijck M, Eichholzer M, Faeh D, Rohrmann S. Ability of a biomarker-based score to predict death from circulatory disease and cancer in NHANES III. Bmc Public Health. 2012;12:895.
9. Lesche D, Sigurdardottir V, Leichtle A, Nakas CT, Christians U, Englberger L, et al. Targeted and global pharmacometabolomics in everolimus-based immunosuppression: association of co-medication and lysophosphatidylcholines with dose requirement. Metabolomics. 2017;14:3.
10. Han H, Jiang X. Overcome Support Vector Machine Diagnosis Overfitting. Cancer Informatics. 2014;13:145–58.
11. Jiang X, Finucane HK, Schumacher FR, Schmit SL, Tyrer JP, Han Y, et al. Shared heritability and functional enrichment across six solid cancers. Nat Commun. 2019;10:431.
12. Mahdavi M, Nassiri M, Kooshyar M, Vakili‐Azghandi M, Avan A, Sandry R, et al. Hereditary breast cancer; Genetic penetrance and current status with BRCA. J Cell Physiol. 2019;234:5741–50.
13. Wendt C, Lindblom A, Arver B, von Wachenfeldt A, Margolin S. Tumour spectrum in non-BRCA hereditary breast cancer families in Sweden. Hered Cancer Clin Pr. 2015;13:15.
14. Snyder C, Hampel H. Hereditary Colorectal Cancer Syndromes. Seminars Oncol Nurs. 2019;
15. Starlinger J, Pallarz S, Ševa J, Rieke D, Sers C, Keilholz U, et al. Variant information systems for precision oncology. Bmc Med Inform Decis. 2018;18:107.
16. Dahlweid F-M, Kämpf M, Leichtle A. Interoperability of laboratory data in Switzerland – a spotlight on Bern. Laboratoriumsmedizin. 2018;42:251–8.
17. Renfro LA, Mallick H, An M-W, Sargent DJ, Mandrekar SJ. Clinical trial designs incorporating predictive biomarkers. Cancer Treat Rev. 2016;43:74 82.
18. Wöckel A, Festl J, Stüber T, Brust K, Stangl S, Heuschmann P, et al. Interdisciplinary Screening, Diagnosis, Therapy and Follow-up of Breast Cancer. Guideline of the DGGG and the DKG (S3-Level, AWMF Registry Number 032/045OL, December 2017) – Part 1 with Recommendations for the Screening, Diagnosis and Therapy of Breast Cancer. Geburtsh Frauenheilk. 2018;78:927–48.
19. Born KB, Levinson W. Choosing Wisely campaigns globally: A shared approach to tackling the problem of overuse in healthcare. J Gen Fam Medicine. 2019;20:9–12.
20. Halfmann M, Stenzhorn H, Gerjets P, Kohlbacher O, Oestermeier U. User-Driven Development of a Novel Molecular Tumor Board Support Tool. In: Sören Auer, Maria-Esther Vidal: Data Integration in the Life Sciences – 13th International Conference, DILS 2018, Hannover, Germany, November 20-21, 2018, Proceedings. Lecture Notes in Computer Science 11371, Springer 2019, ISBN 978-3-030-06015-2
21. Ozanne EM, Howe R, Mallinson D, Esserman L, Veer LJ, Kaplan CP. Evaluation of National Comprehensive Cancer Network guideline‐based Tool for Risk Assessment for breast and ovarian Cancer (N‐TRAC): A patient‐reported survey for genetic high‐risk assessment for breast and ovarian cancers in women. J Genet Couns. 2019;
22. Leichtle A, Ceglarek U, Weinert P, Nakas CT, Nuoffer J-M, Kase J, et al. Pancreatic carcinoma, pancreatitis, and healthy controls: metabolite models in a three-class diagnostic dilemma. Metabolomics. 2013;9:677–87.
23. de la Torre F, Campoy E. Diagnostic role of tumor markers. Medicina Clínica Engl Ed. 2019;
24. Hori SS, Gambhir SS. Mathematical Model Identifies Blood Biomarker–Based Early Cancer Detection Strategies and Limitations. Sci Transl Med. 2011;3:109ra116-109ra116.
25. Wolter C, Luppa P, Breul J, Fink U, Hanauske A-R, Präuer H, et al. Humorale Tumormarker. Deutsches Ärzteblatt. 1996;93:3346–53.
26. Konforte D, Diamandis E. Is Early Detection of Cancer with Circulating Biomarkers Feasible? Clin Chem. 2012;59.
27. Alimirzaie S, Bagherzadeh M, Akbari MR. Liquid Biopsy in Breast Cancer: A Comprehensive Review. Clin Genet. 2019;
28. van der Toom EE, Verdone JE, Gorin MA, Pienta KJ. Technical challenges in the isolation and analysis of circulating tumor cells. Oncotarget. 2016;7:62754–66.
29. Simon R. Sensitivity, Specificity, PPV, and NPV for Predictive Biomarkers. Jnci J National Cancer Inst. 2015;107:djv153.
30. Vickers AJ. Decision Analysis for the Evaluation of Diagnostic Tests, Prediction Models, and Molecular Markers. Am Statistician. 2008;62:314–20.
31. Stieber P, Heinemann V. Sinnvoller Einsatz von Tumormarkern / Sensible use of tumor markers. Laboratoriumsmedizin. 2008;32:339–60.
32. Lund F, Petersen P, Pedersen M, Hassan S, Sölétormos G. Criteria to interpret cancer biomarker increments crossing the recommended cut-off compared in a simulation model focusing on false positive signals and tumour detection time. Clin Chim Acta. 2014;431:192–7.
33. Petersen P, Sölétormos G, Pedersen M, Lund F. Interpretation of increments in serial tumour biomarker concentrations depends on the distance of the baseline concentration from the cut-off. Clin Chem Lab Med. 2011;49:303–10.
34. Passerini R, Cassatella MC, Boveri S, Salvatici M, Radice D, Zorzino L, et al. The Pitfalls of CA19-9Routine Testing and Comparison of Two Automated Immunoassays in a Reference Oncology Center. Am J Clin Pathol. 2012;138:281–7.
35. Febbo PG, Ladanyi M, Aldape KD, Marzo A, Hammond EM, Hayes DF, et al. NCCN Task Force Report: Evaluating the Clinical Utility of Tumor Markers in Oncology. J Natl Compr Canc Ne. 2011;9:S-1-S-32.
36. Smith JS, Alderete B, Minn Y, Borell TJ, Perry A, Mohapatra G, et al. Localization of common deletion regions on 1p and 19q in human gliomas and their association with histological subtype. Oncogene. 1999;18:1202759.
37. Mok TS, Wu Y-L, Thongprasert S, Yang C-H, Chu D-T, Saijo N, et al. Gefitinib or Carboplatin–Paclitaxel in Pulmonary Adenocarcinoma. New Engl J Medicine. 2009;361:947–57.
38. Schröder FH, Hugosson J, Roobol MJ, Tammela TL, Ciatto S, Nelen V, et al. Screening and Prostate-Cancer Mortality in a Randomized European Study. New Engl J Medicine. 2009;360:1320–8.
39. Duffy MJ. Tumor Markers in Clinical Practice: A Review Focusing on Common Solid Cancers. Med Prin Pract. 2012;22:4–11.
40. Sharma S. Tumor markers in clinical practice: General principles and guidelines. Indian J Medical Paediatr Oncol. 2009;30:1–8.
41. Handy B. The Clinical Utility of Tumor Markers. Lab Med. 2009;40:99–103.
42. Sturgeon C, Duffy MJ, Stenman U-H, Lilja H, Brünner N, Chan DW, et al. National Academy of Clinical Biochemistry Laboratory Medicine Practice Guidelines for Use of Tumor Markers in Testicular, Prostate, Colorectal, Breast, and Ovarian Cancers. Clin Chem. 2008;54:e11–79.
43. Jørgensen J. Clinical application of companion diagnostics. Trends Mol Med. 2015;21:405–7.
44. Manceau H, Amrani K, Peoc’h K. Personalized medicine, pharmacogenomic and companion biomarker. Ann Biol Clin-paris. 2017;75:631–6.
45. Seo M, Cairns J. Do cancer biomarkers make targeted therapies cost-effective? A systematic review in metastatic colorectal cancer. Plos One. 2018;13:e0204496.
46. Meng Q, Shi S, Liang C, Liang D, Xu W, Ji S, et al. Diagnostic and prognostic value of carcinoembryonic antigen in pancreatic cancer: a systematic review and meta-analysis. Oncotargets Ther. 2017;10:4591–8.
47. Nahm CB, Turchini J, Jamieson N, Moon E, Sioson L, Itchins M, et al. Biomarker panel predicts survival after resection in pancreatic ductal adenocarcinoma: A multi-institutional cohort study. European J Surg Oncol J European Soc Surg Oncol Br Assoc Surg Oncol. 2018;45:218–24.
48. Torén W, Ansari D, Andersson R. Immunohistochemical investigation of prognostic biomarkers in resected colorectal liver metastases: a systematic review and meta-analysis. Cancer Cell Int. 2018;18:217.
49. Huang X, Zhang W, Shao Z. Prognostic and diagnostic significance of circRNAs expression in hepatocellular carcinoma patients: A meta‐analysis. Cancer Med-us. 2019;
50. Ding W, Chen G, Shi T. Integrative analysis identifies potential DNA methylation biomarkers for pan-cancer diagnosis and prognosis. Epigenetics. 2019;1–14.
info@onco-suisse
- Vol. 9
- Ausgabe 1
- Februar 2019